Hieff NGS® C112P1 Fast Tagment DNA Library Prep Kit for Illumina® (for 1 ng)/ 1 ng 转座酶建库试剂盒
产品介绍
Hieff NGS® C112P1 Fast Tagment DNA Library Prep Kit for Illumina® (for1 ng)是针对 lllumina®高通量测序平台专业开发设计的转座酶法建库试剂盒,适用于1 ng的基因组 DNA、cDNA、扩增子(>500 bp)等样本的建库。与常规分步法建库相比,Hieff NGS® C112P1 Fast Tagment DNA Library Prep Kit for Illumina®采用新型转座酶和优化的缓冲体系,仅需10 min即可完成DNA片段化、末端修复和接头连接过程,显著缩短建库时间至1.5小时以内,并获得优异的测序质量。此外,本试剂盒已经不同GC含量DNA样本的验证,无偏好性或仅具有极低的偏好性。本试剂盒提供的所有试剂盒组分,都经过严格的质量与功能验证,最高程度保障产品优异性能与批间稳定性。
产品特色
- 建库时间短:仅需10 min即可完成DNA片段化、末端修复和接头连接过程,显著缩短建库时间至1.5小时以内。
- 严格背景菌控制:专人操作,车间洁净,全流程监控,质控严格。
- 保证批次稳定性:有试剂盒组分,都经过严格的质量与功能验证,最高程度保障产品优异性能与批间稳定性。
应用案例
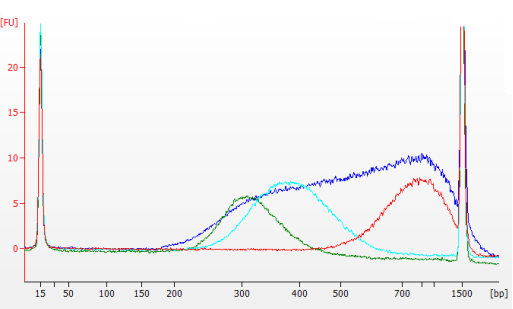
使用 Hieff NGS® Fast Tagment DNA Library Prep Kit for Illumina®对1 ng嗜盐杆菌gDNA样本建库,并分别使用1.0×磁珠进行纯化,0.8×/0.2×、0.7×/0.2×、0.5×/0.15×磁珠比例进行长度分选,结果使用Agilent 2100 Bioanalyzer进行检测。
存储条件
-25~-15℃保存。
COA
相关产品
联系我们