Hieff NGS® OnePot Pro DNA Library Prep Kit V3/ 兼容Illumina和MGI双平台酶切DNA建库试剂盒V3
产品介绍
Hieff NGS® OnePot Pro DNA Library Prep Kit V3 是一款可用于Illumina®和MGI®高通量测序平台的新一代酶切法建库试剂盒。与传统的建库法比较,本品采用高质量的片段化酶,摆脱了繁琐的超声过程。将片段化模块与末端修复模块合二为一,极大的降低了建库的时间和成本、连接模块的酶和buffer预混,简化了操作流程,极大地降低了建库的时间和成本,更加适合于自动化建库。本试剂盒具有更高的文库转化率,可应用于常规动植物基因组、微生物基因组等样本,同时能兼容FFPE DNA样本的建库。在前一代建库试剂盒的基础上,改进了片段化/末修/加A模块,降低了试剂对样本的GC偏好性,提高了末修和加dA尾的效率,提高了试剂的稳定性;本试剂盒使用了最新优化的连接酶,改善了接头连接效率。同时,本试剂盒可搭配Illumina®或MGI®的接头和Primer,用于Illumina®和MGI®高通量测序平台测序。
产品特色
- 适用1 ng - 1 μg的基因组DNA、全长cDNA等样本。
- 高质量片段化酶,可随机切割双链DNA,酶切片段偏好性低。
- 片段化、末端修复/加A一步完成。
- 强扩增效率的高保真酶,显著提高文库质量及产量。
- 适用于FFPE DNA样本。
- 严格的批次性能与稳定性质控。
应用案例
1、较高的文库产出
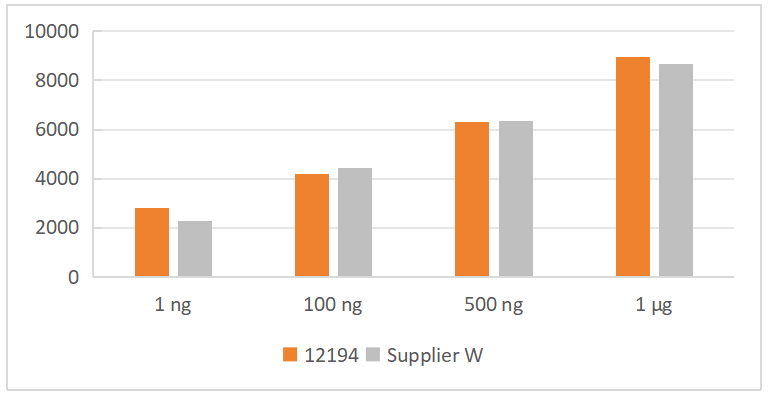
Human gDNA为模板,以不同的样本投入量,12194与竞品W试剂盒相比,12194的文库产量与竞品相当。数据显示不同投入量文库产量对比,12194有高效的文库转化效率。
2、极低的假阳性率
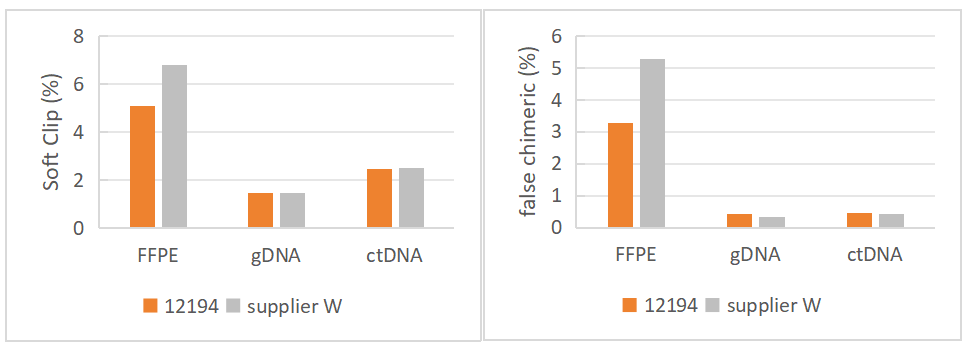
用12194酶法片段化建库试剂盒和竞品W对3种标准品样本(FFPE标准品、gDNA标准品、ctDNA标准品)建库,数据显示,无论是FFPE样本、gDNA、还是ctDNA,12194的Soft Clip、false chimeric都低于或近似于竞品W。
存储条件
-25~-15℃保存。
COA
相关产品
联系我们