Q:机械法建库试剂盒可用于哪些应用?
A:全基因组测序,靶向捕获测序,扩增子测序,ChIP-Seq 等。
Q:机械法建库试剂盒可用于哪些样本?
A:常规动植物基因组 微生物基因组 FFPE cfDNA ChIP DNA 等。
Q:初始DNA 投入量范围是多少?
A:500 pg-1 μg Fragment DNA 样本。12201 适用于 500 pg-500 ng Fragment DNA 样本
Q:12199 12200 12201 建库试剂盒建库接头搭配选择?
A:根据客户需求提供。如完整接头(Cat#12615-12618):单端 48 index(如客户需求更多 index 可咨询技术);单端短接头(Cat#12611-12612):单端 96 index;双端短接头(Cat#12412-12413): 双端 384 index;UDI 短接头(Cat#12404-12407):384 双端唯一Index
Q:我们的建库试剂盒里面包含接头吗?
A:我们所有建库试剂盒都是不包含接头的,接头有设置单独的货号,可根据客户选择提供。
Q:12199 12200 12201 扩增模块如何选择?
A:翌圣提供两种扩增模块。高扩增效率模块(Cat#12620)注重高效扩增,文库产量高但保真度受影 响;高保真模块(Cat#12621/12624)注重高保真扩增但文库产量受影响,适用于后续 SNP 检测。目前客户如果需要高产量扩增,推荐 12199 Ultima 建库试剂盒;如客户后续做 SNP 检测相关,可推荐12200 MaxUpⅡ DNA/12201 建库试剂盒。
Q:12199 12200 12201 13310 13311 纯化磁珠如何选择?
A:Cat#12601,Hieff NGS DNA Selection Beads 或Cat#A63880,AMPure XP Beads 或其他等效产品。
Q:12199 12200 建库一次需要多少磁珠?
A: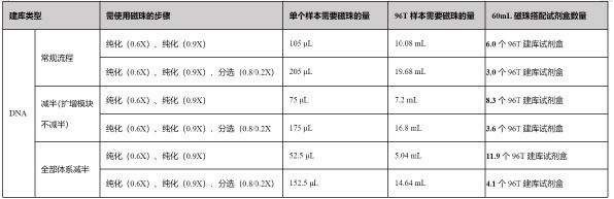
Q:建库试剂盒本身不会造成文库锯齿状,更多应考虑样本和实验过程问题:
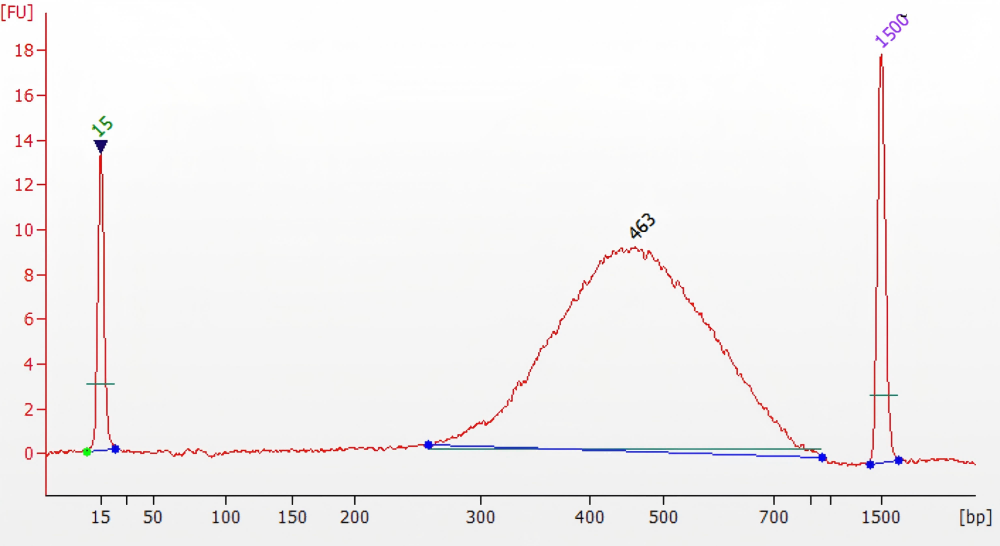
A:1)Input DNA 量偏低。根据maxup II 说明书推荐,全基因组测序Input DNA 应大于 50ng,靶向捕获测序Input DNA 应大于 10ng,目前客户的上样量偏低(1ng,10ng 和 50ng),可能造成建库不成功。 2)样本片段化不完全。部分酶法片段化方式,可能造成锯齿状。 3)PCR 扩增循环数过高。根据经验,当 cycle 大于 15 个循环时,可能造成非均一扩增,呈现文库锯齿状。
Q:文库扩增的效率和保真性,怎么从这个图中看扩增效率和保真性?
A:扩增效率即扩增一定产量所用的 PCR 循环数,相同产量PCR 循环数越低则扩增效率越高;保真性即 PCR 扩增时候出现碱基错配的概率,错配率越低则保真性越高。以 CanaceⅡ高保真酶为例,保真性是普通Taq 酶的 52 倍,是 Pfu 的 6 倍。高保真酶扩增的时候必来带来一定的偏好性,会优先扩增中等GC 含量的片段,下图即体现的是高保真酶的偏好性问题。正常情况下,若无 GC 偏好性,也就是说所有的片段经过相同的扩增之后都是一样的。这时候蓝色的圆圈点也就是与纵坐标1.0 直线重合。如果蓝色圆圈在纵坐标 1.0 以上,则可能是出现对于该处 GC 含量的偏好性扩增导致。
Q:不同测序深度Coverage 解读?
A:Coverage 即基因组覆盖度,指的是测序获得的序列占整个基因组的比例。不同测序深度下序列的coverage 肯定也是有差异的。简单来说,10×意思就是说有 10 个测序 reads map 到基因组的某个位置,200×是说有 200 个测序 reads map 到基因组的这个位置。一般情况下测序深度越高,coverage 也会随之降低。
Q:构建得到的文库产量低
A: 1. DNA 质量低,样本中含抑制物(抑制酶活性等) Input DNA 制备过程中带入的高浓度金属离子螯合剂或者其他盐,可能会影响末端修复以及 A 尾添加等步骤的反应效率,DNA 在TE 溶液中进行片段化,不要在无菌超纯水中进行。 2.RNA 污染 样本中存在RNA 污染会导致DNA 定量不准确,使文库构建时DNA 上样量不足。 3.PCR free 的时候上样量不足 当使用完整接头且Input DNA 大于 200 ng 时候,不用 PCR 扩增。如果文库拷贝数过低不能进行直 接测序,可在接头连接完成后对DNA 文库进行PCR 扩增。 4.接头出现降解:接头存储不当会出现降解,影响连接效率。一般稀释过的接头可在室温放置 48h。 判断接头是否降解的方法: 用安捷伦 2100 毛细血管电泳检测,通过电泳条带大小判断;如果未发生降解,则条带大小大概为 62bp(complete adapter),如果发生降解,则无条带或条带很小。 用Qubit 酶标仪等检测仪器进行浓度测量,粗略判断接头是否降解。 检测方法:取 1uL,15uM 的complete 接头(12615ES-12618ES)若 Qubit 检测浓度 600ng/ul 左右,则 接头正常,反之若小于 600ng/ul,则接头可能降解成单链或小片段。酶标仪检测同理。
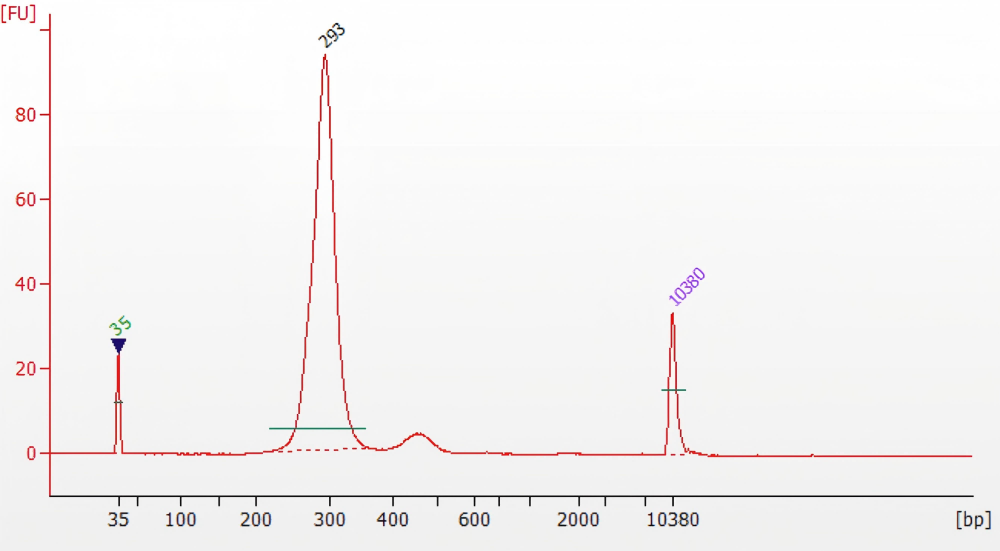
Q:文库电泳的时候条带异常
A:1 出现小片段(60-120bp) 小片段一般为接头片段或接头所形成的二聚体,一般可用 12601 DNA 磁珠进行纯化; 2.文库富集之后出现大片段 一般文库接头在 120 bp 左右,若文库片段增加幅度超过了 120bp 则可能是扩增过度,导致片段扩增异常。此时需减少PCR 循环数 3.接头连接前片段异常片段化时间或者条件选择不当。