Hieff NGS® Dual Barcode Fast-Pace DNA Cyclization Kit for MGI/双端barcode MGI环化试剂
产品介绍
Hieff NGS® Dual Barcode Fast-Pace DNA Cyclization Kit for MGI®是针对MGI高通量测序平台专门开发设计的单链环化试剂盒。本产品采用高质量的酶和经优化后的Buffer组成,显著提高反应效率,使整个环化消化流程在30 min内完成。本试剂盒适用于所有连接华大标准的双标签PCR接头文库扩增产物,除建库试剂本身的限制外,本环化试剂盒不限MGI测序平台。
产品特色
高效:有效环化率高
快速:环化用时缩短至1h
应用案例
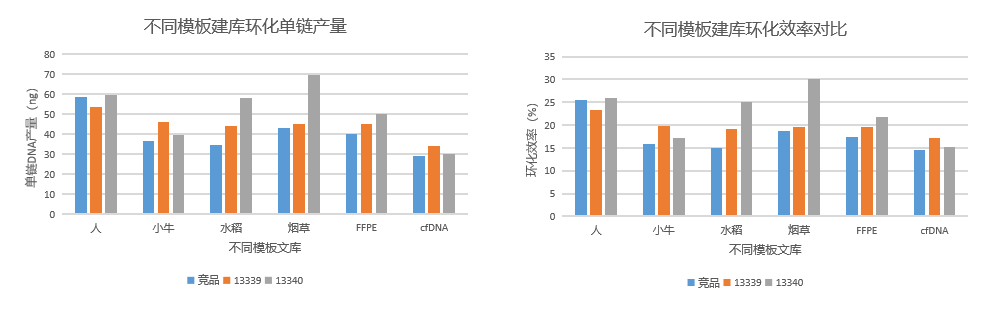
不同模板建库环化与竞品对比
存储条件
-25~-15℃保存,有效期1年。
COA