Hieff NGS® Ultima DNA Library Prep Kit for Illumina®是针对Illumina®高通量测序平台定向优化而成的新一代建库试剂盒。作为全新的升级版本,本产品采用高质量的酶学组成,简化的操作流程,可显著提高低质量样本文库转化率与扩增效率,具有广泛的样本适应性,同时兼容FFPE、cfDNA、ChIP DNA等样本,助力获得优异的测序数据。
- 适用500pg-1μgDNA样本。
- 兼容cfDNA、FFPE等低质量样本。
- 高效的文库转化率与扩增效率。
- 多样本验证可获得优异的文库与测序数据。
- 严格的批次性能与稳定性质控。
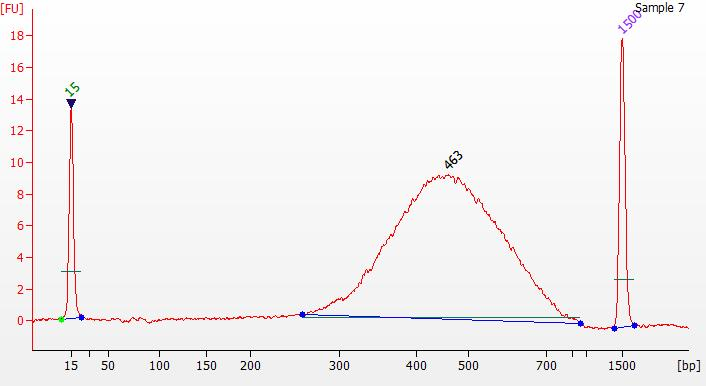
Ultima DNA 建库试剂盒用于 FFPE 样本建库(未分选)
-25~-15℃保存。
Q:12197与12199有什么区别?
A:12197建库起始量是100 pg-1 μg,12199建库起始量是500 pg-1 μg。
Q:建库试剂盒里面包含接头吗?
A:我们所有建库试剂盒都是不包含接头的,接头有设置单独的货号,可根据客户选择提供。
Q:接头连接之后可以直接进行分选吗?
A:不可以的,Ligation Enhancer 内含高浓度的PEG,会对分选造成显著影响,所以接头连接后如果进行
片段分选前必须先纯化。
Q:构建得到的文库产量偏低
A:(1)模板DNA 质量低,样本中含抑制物(抑制酶活性等)影响后续的反应效率,可以适当提高循环数或更换高质量模板;
(2)RNA 污染,样本中存在RNA污染会导致DNA定量不准确,使文库构建时DNA起始量不足;
(3)DNA起始量不足,可重新确定模板浓度是否准确。
(4)接头出现降解:接头存储不当会出现降解,影响连接效率。一般稀释过的接头可在室温放置 48h,判断接头是否降解的方法:
a. 用安捷伦 2100 毛细血管电泳检测,通过电泳条带大小判断;如果未发生降解,则条带大小大概为 62bp(complete adapter),如果发生降解,则无条带或条带很小。
b.用Qubit 酶标仪等检测仪器进行浓度测量,粗略判断接头是否降解。
检测方法:取 1uL,15uM 的complete 接头(12615ES-12618ES)若 Qubit 检测浓度 600ng/ul 左右,则
接头正常,反之若小于 600ng/ul,则接头可能降解成单链或小片段。酶标仪检测同理。
Q:文库电泳的时候条带异常:二聚体污染,小片段或大片段残留,文库大小偏大或偏小、宽峰、锯齿状峰形
A:(1)二聚体污染:包括接头二聚体(<120bp)和引物二聚体(<100bp),一般可用 12601 DNA 磁珠纯化后去除,接头二聚体明显过多的话可以降低接头用量。
(2)文库富集之后出现过多小片段和大片段:片段化条件不合适,文库磁珠分选比例不正确,PCR循环数太高。
(3)文库大小偏大或偏小:片段化条件或磁珠分选比例不合适,偏小的可能是过度片段化,。
(4)宽峰:a.分选体系问题,如因一轮分选方案中磁珠用量过少,可调整一下分选体系、增加或减少一轮分选中的磁珠体积,使主峰的位置在合格的上机文库范围内;b.样本DNA质量不好,发生降解,条带弥散。
(5)非正常的锯齿状峰形:a.Input DNA 量偏低,适当增加DNA起始量;b.样本片段化不完全,部分酶法片段化方式,可能造成锯齿状;c. PCR 扩增循环数过高,根据经验,当 cycle 大于 15 个循环时,可能造成非均一扩增,呈现文库锯齿状。
[1] Feng YL, Liu Q, Chen RD, et al. DNA nicks induce mutational signatures associated with BRCA1 deficiency. Nat Commun. 2022;13(1):4285. Published 2022 Jul 25. doi:10.1038/s41467-022-32011-x(IF:17.694)
