CUT&Tag试剂盒——看ChIP-Seq的进阶如何玩转DNA蛋白质互作研究!
表观遗传是指DNA序列不发生变化但基因表达却发生了可遗传的改变,即基因型未发生变化而表型却发生了改变。表观遗传调控是指基因的表达水平受各种修饰的影响,其分子基础有两个方面,即针对DNA本身的修饰和对组蛋白的修饰。蛋白质与DNA相互作用是基因转录调控的关键,也是基因转录启动的前提。传统蛋白质DNA互作研究常用的有凝胶阻滞,酵母杂交,ChIP-Seq等,其中ChIP-Seq可以完整的反映与靶蛋白结合的DNA序列,是蛋白质与DNA互作的经典方法。
但是近年来CUT&Tag(Cleavage Under Targets and Tagment)作为一种新兴的DNA蛋白互作研究技术,凭借其信噪比高,可重复性好,实验周期更快等优势,在植物以及动物细胞中的成功应用,相关科研成果也陆续发表[1-2],受到广大科研工作者的信赖。

2009年,Dominic Schmidt等将ChIp与当时热门技术NGS结合,成就了ChIP-Seq技术,从而能够在全基因组范围内检测与蛋白相互作用的DNA区域。
1.ChIP-Seq
1) 实验原理
甲醛处理细胞使目标蛋白与DNA交联,通过超声波将交联后的染色质打断成小片段,一般在200-600 bp范围内,再利用抗原抗体的特异性识别反应,将与目的蛋白相结合的DNA片段沉淀下来,纯化之后进行文库的构建并测序。
2)实验流程
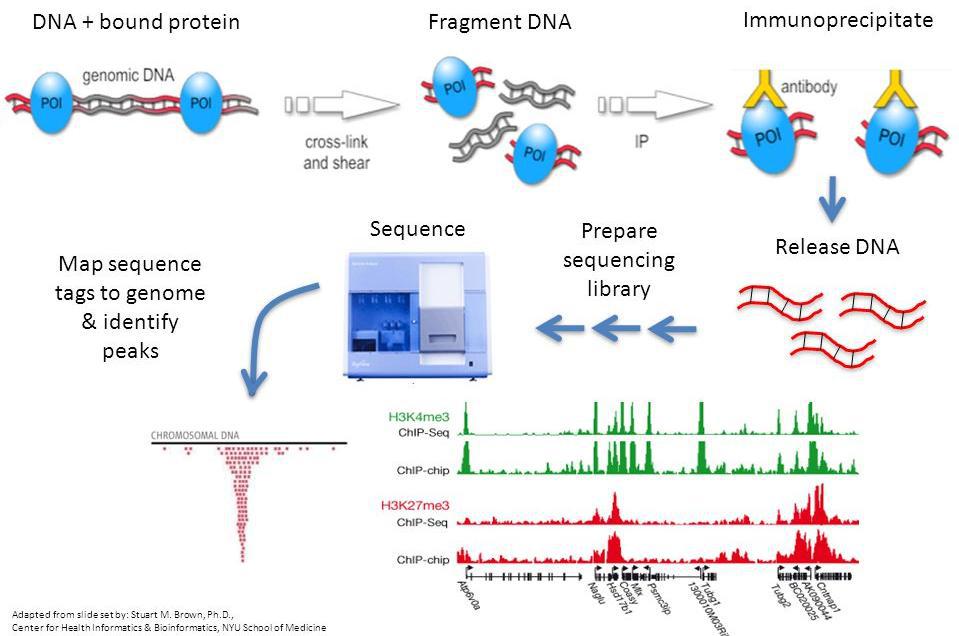
图1. ChIP-Seq实验流程
3)实验难点
» 时间长,一次实验需要2~3天,且操作误差对实验结果影响大;
» 起始的细胞需求量大,需要107个细胞,且背景噪音较高;
» 甲醛交联与免疫共沉淀需根据经验控制时间,会造成非特异结合增加;
» 对抗体的特异性要求高,需要ChIP级别的抗体。
2019年,Henikoff实验室发明了CUT&Tag,使用ProteinA/G-Tn5从而简化了二代测序的步骤,并且极大降低了细胞的起始数量。
2.CUT&Tag
1)实验原理
CUT&Tag是蛋白质与DNA互作的革新技术,无需通过甲醛交联以及免疫共沉淀,其核心技术为pA/G-Tn5 Transposase,将Protein A/G与Tn5转座酶进行融合,使得与抗体结合的同时,Tn5切割核小体上缠绕的DNA片段,获得目的序列。
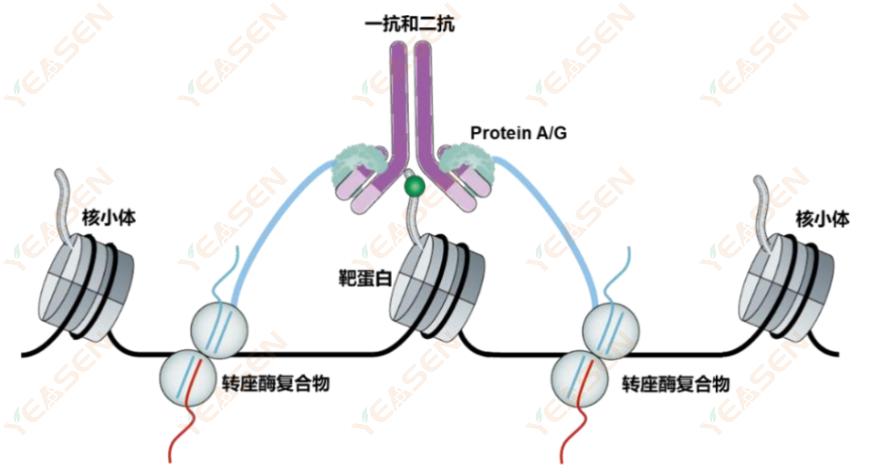
图2. CUT&Tag原理图
2)实验流程
利用连有刀豆蛋白A的磁珠(ConA)结合细胞(与细胞膜上的糖蛋白结合)并利用洋地黄皂苷进行细胞膜通透,接着孵育靶蛋白的抗体,二抗以及Protein A/G Tn5。抗体和Protein A/G与Tn5能够进入细胞核,Tn5此时在Protein A/G的帮助下切割靶蛋白附近的DNA序列,由于Tn5是经过修饰的,切割的同时会在片段的两端加上一部分Adapter序列,再通过PCR文库扩增可得到对应的文库进行测序。
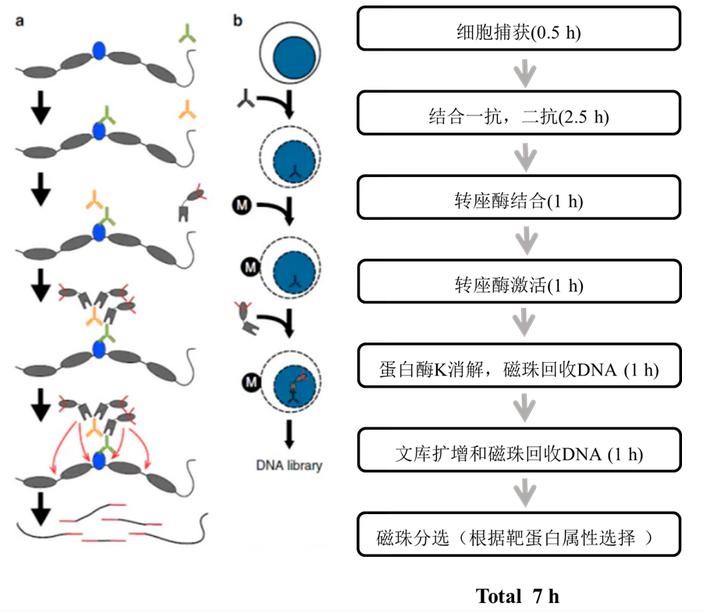
图3. CUT&Tag实验流程
3)技术优势
» 流程简便:无需甲醛交联、超声打断、免疫共沉淀,无需补平,加A和接头;
» 可视化操作:利用ConA beads结合细胞,操作可视化;
» 磁珠回收:片段化后DNA磁珠回收基因组DNA,简化操作,减少杂质引入;
» 特异性好:抗体与靶蛋白特异性结合进行目标区域定位;
» 起始量低:所需细胞数10^7下降到10^2,甚至可以做单细胞水平研究;
» 性价比高:无需ChIP级别抗体,背景噪音低,所需测序深度少;
» 重复性好:实验重复结果一致性好。
Henikoff等通过三种方法来对组蛋白H3K27me3进行研究,在相同的数据量(8M Reads)情况下,三种方法的染色体景观相似,但ChIP-Seq的背景较高,CUT&Tag的信号更强,信噪比更高。
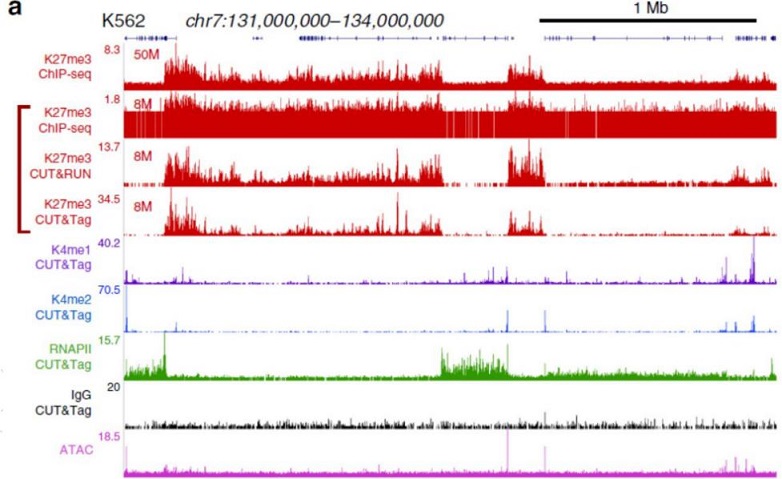
图4. CUT&Tag、CUT&RUN(Henikoff于2017年发明了新技术)的peaks鉴定结果与ChIP-Seq的对比(Henikoff,2019)
同时CUT&Tag可以对极少量细胞(60个细胞)进行操作,实现单细胞测序。Protein A-Tn5在细胞内进行核酸切割,PCR之前的反应都在细胞内进行。Henikoff博士通过分配单个细胞到5184纳米孔,再加入带随机标签的引物进行扩增的办法,实现单细胞测序。
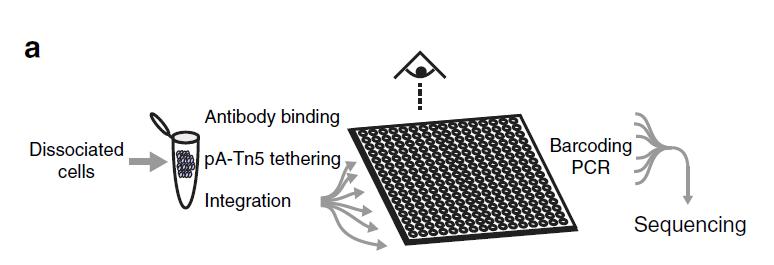
图5. 单细胞CUT&Tag测序流程(Henikoff,2019)
翌圣生物CUT&Tag试剂盒相较于传统的ChIP-seq,优化了反应体系和实验操作流程,文库时间更短(仅需7小时),起始样本要求更低,抗体投入量更少,文库产量更高等优点,是蛋白质-DNA互作研究技术的又一颠覆性创新。
1.更高的文库质量。
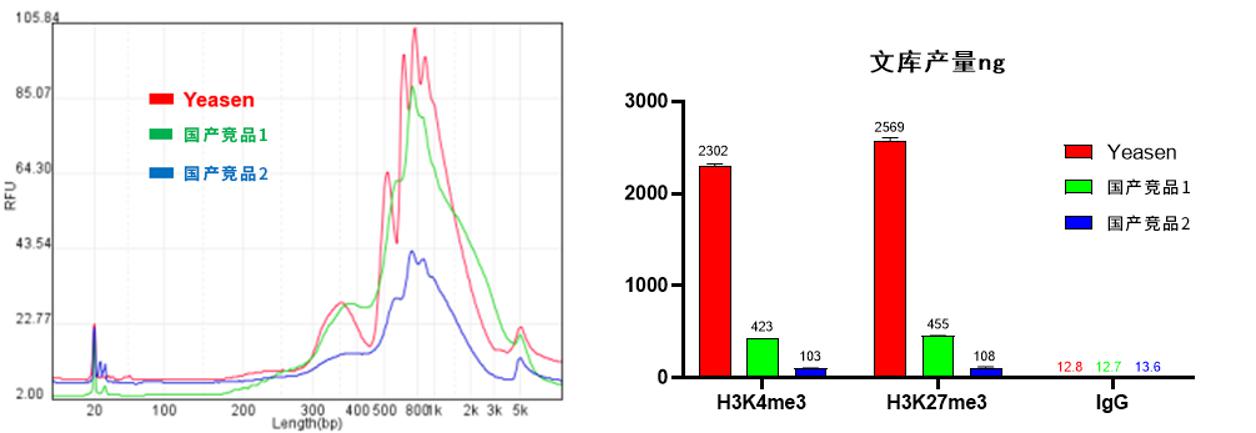
图6. 文库质检图
2.数据质量好。
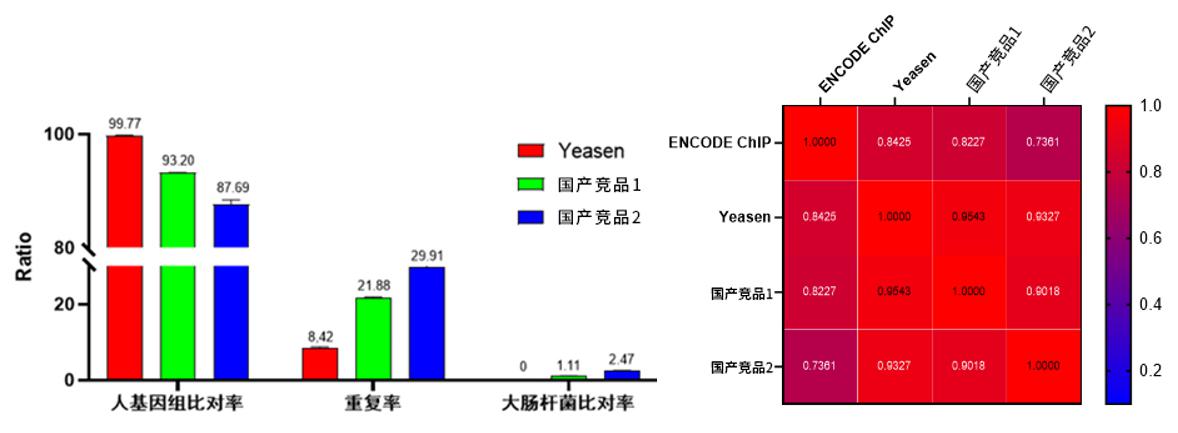
图7.测序数据对比图
3.更高的信噪比
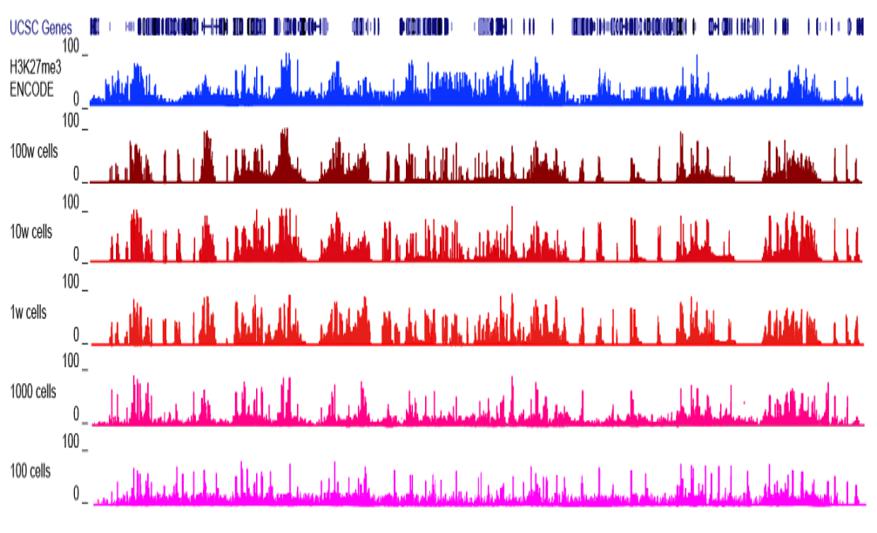
图8.信噪比展示
|
产品名称 |
产品编号 |
规格 |
|
Hieff NGS® In-Situ DNA Binding Profiling Library Prep Kit for Illumina® V2 |
12597ES12/48 |
12 T/48 T |
|
12592ES12/48 |
12 T/48 T |
|
|
12416ES96 |
96 index |
[1]. Dan Lu, Liu L, Sun Y, Song J, Yin Q, Zhang G, Qi F, Hu Z, Yang Z, Zhou Z, Hu Y, Zhang L, Ji J, Zhao X, Jin Y, McNutt MA, Yin Y. The phosphatase PAC1 acts as a T cell suppressor and attenuates host antitumor immunity. Nat Immunol. 2020 Mar;21(3):287-297. doi: 10.1038/s41590-019-0577-9. Epub 2020 Jan 13. PMID: 31932812.
[2]. Tao X, Feng S, Zhao T, Guan X. Efficient chromatin profiling of H3K4me3 modification in cotton using CUT&Tag. Plant Methods. 2020 Aug 31;16:120. doi: 10.1186/s13007-020-00664-8. PMID: 32884577; PMCID: PMC7460760.
[3]. Kaya-Okur, H. S., Wu, S. J., Codomo, C. A., Pledger, E. S., Bryson, T. D., Henikoff, J. G., ... & Henikoff, S. (2019). CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature communications, 10(1), 1930.
HB210802
推荐商品
