NGS二代测序相关技术知多少
NGS,或者是二代测序(当然还有三代测序、四代测序)这几个词,相信大家就算未接触过,也已经听过,NGS到底是做什么呢?或者说,NGS到底研究的是什么?在这里,我们来详细讲解讲解吧。
二代测序相关的技术可以分为三大类,DNA研究相关技术、RNA研究相关技术和表观研究相关技术(我们主要来给大家讲述下二代测序相关的技术,谁让目前属二代测序相关技术及应用最多呢)。
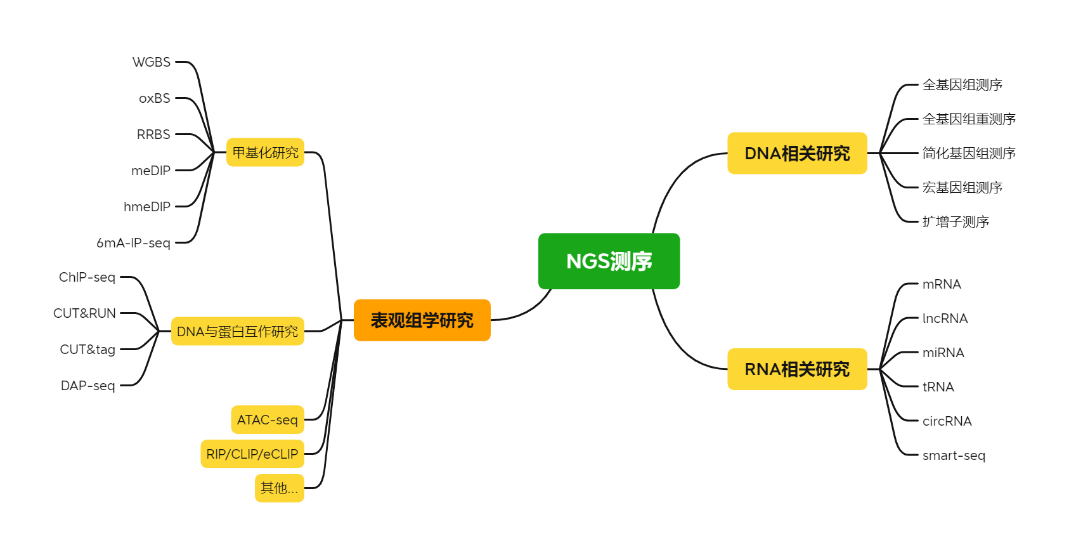
图:NGS测序图鉴
1、DNA研究相关技术
DNA相关研究技术主要步骤离不开:DNA提取、DNA片段化、DNA文库构建、测序和数据分析。
看起来,DNA相关研究技术操作步骤似乎没有那么难,那么,如果你觉得DNA研究很简单的话,那你就真是too young too simple啦。
首先,DNA研究相关技术里,全基因组测序WGS是逃脱不开的技术,从人类基因组计划实施完成之后,人类开始进入了基因组时代,不同物种的基因组信息被相继报道,关于基因组的生信分析手段也层出不穷,要来讲基因组的故事,可能得讲述个“两三百年”了,所以这里,我们就不去赘述它。但是大家要知道,所有的关于二代测序研究,在进入这个领域之前,我们还是要去确认下所要研究的物种是否有基因组,当然了,这里可能会有些杠精默默吐槽,无参分析现在都可以做的。嗯,我知道啊,但是无参分析从来也只是不得已而为之,在有参考序列时,尽量选择用有参的方式进行数据分析。
在基因组测序上,还有全基因组重测序和简化基因组测序,这类技术目前主要应用与构建遗传图谱、种质资源鉴定、动植物遗传育种、结构变异检测、性状基因定位和群体进化分析等,衍生了一些分析的专有名称如BSA、SNP、Indel、SV、QTL、GWAS等,都是在根据不同的样本数量及分析需求进行调整。除此之外,还有一些做环境样本如土壤、粪便、肠道和水体等或者病原样本的宏基因组的,也是基于单个样本基因组研究技术发展起来的。当然,还有一些基于群体的但是研究的是某个或者某些基因部分序列的技术如DNA条形码、宏条形码等或者是16S/18S/ITS测序、功能基因组测序、食性分析等扩增子测序。
|
产品定位 |
产品名称 |
产品货号 |
规格 |
|
机械法建库 |
12199ES24/96 |
24/96 T |
|
|
12201ES24/96 |
24/96 T |
||
|
酶切法建库 |
12205ES24/96 |
24/96 T |
|
|
机械法建库 |
13311ES16/96 |
16/96 T |
|
|
酶切法建库 |
13322ES16/96 |
16/96 T |
2、RNA研究相关技术
RNA相关研究技术的主要步骤也是一样,离不开RNA提取、mRNA捕获(rRNA去除)、RNA文库构建、测序和数据分析。
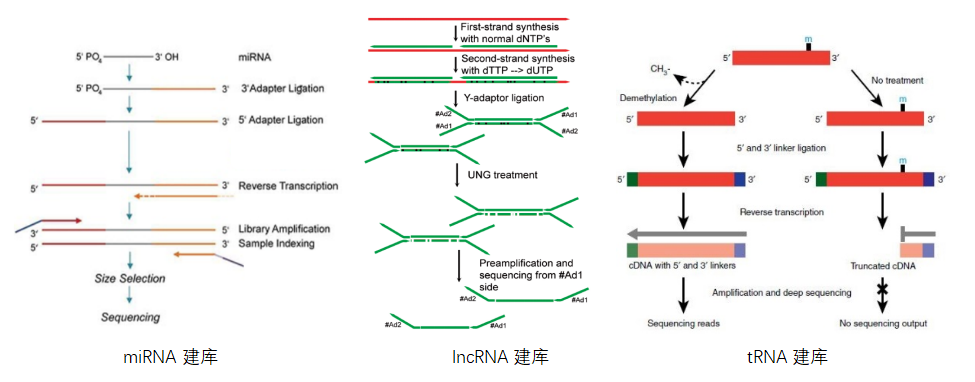
RNA研究目前主要的研究对象是mRNA、miRNA、circRNA、lncRNA以及tRNA。根据研究对象的特性,建库的方法也不相同。比如mRNA有polyA尾,所以使用oligo dT beads捕获ployA;而miRNA因为片段较小,通常用非变性胶跑胶检测后切胶回收,circRNA建库分两种情况,一种是只构建circRNA的文库,这种时候,需要先去除线性化的RNA,再来构建circRNA的文库,当然,还有另外一种就是用去除rRNA的方式构建lncRNA文库,这样既能够得到mRNA信息、lncRNA的信息,还能得到部分circRNA;研究tRNA呢,得特异性捕获tRNA得发夹结构,然后加上去甲基化得步骤,之后构建tRNA文库。
在这里,要来讲一个特殊的转录组研究,就是单细胞RNA-seq,单个细胞因为RNA量少,使用常规的建库方法通常容易失败。这时,我们需要让整个实验尽量在一管中实验,而不是常规的需要在整个实验流程中需要不断的更换EP管。目前,大多采用的是磁珠来实现一管操作。因为建库时,通常构建的是mRNA文库。
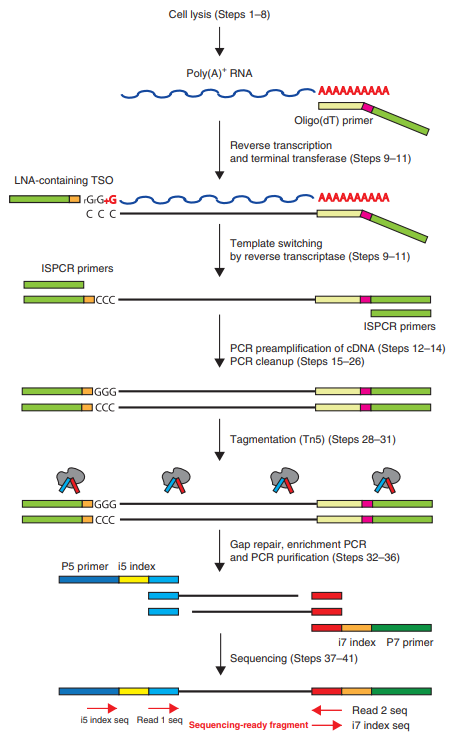
Smart-seq2实验流程【1】
|
产品定位 |
产品名称 |
产品编号 |
规格 |
|
mRNA常规文库和RNA链特异文库 |
Hieff NGS® Ultima Dual-mode mRNA Library Prep Kit for Illumina® 双模式mRNA建库试剂盒 |
12301ES24/96 |
24/96 T |
|
Hieff NGS® Ultima Dual-mode mRNA Library Prep Kit for MGI® 双模式mRNA 建库试剂盒 |
13330ES24/96 |
24/96 T |
|
|
LncRNA+mRNA常规文库及链特异文库 |
Hieff NGS® Ultima Dual-mode RNA Library Prep Kit for Illumina® |
12252ES24/96 |
24/96 T |
|
13333ES24/96 |
24/96 T |
||
|
一步法RNA建库试剂盒(含人rRNA去除试剂) |
Hieff NGS® Fast RNA Library Prep Kit for Illumina® 极速RNA 建库试剂盒 |
12304ES24/96 |
24/96 T |
|
单细胞RNA文库构建 |
Hieff NGS® Single Cell/Low Input cDNA Synthesis & Amplification Module |
12500ES24/96 |
24/96 T |
|
Hieff NGS® Fast Tagment DNA Library Prep Kit for Illumina® (for 1 ng) |
12206ES24/96 |
24/96 T |
3、表观组学相关技术
表观组学主要研究的是核酸不发生变化,但是表型发生变化的机制。目前表观组学研究分为四大部分:甲基化研究、组蛋白修饰研究、染色质重塑研究以及非编码RNA调控研究。而这中间涉及的技术类型非常的多。
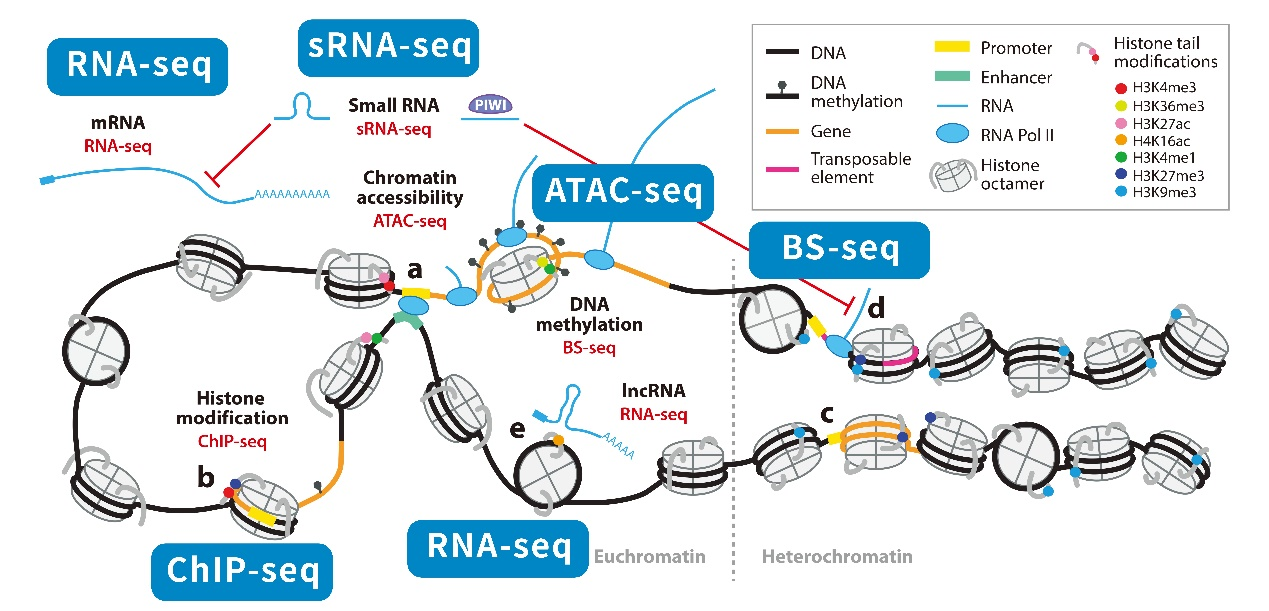
图:表观遗传调控图鉴
3.1 甲基化研究
甲基化研究分两大类,一类是DNA甲基化,一类是RNA甲基化。DNA甲基化目前研究的是5mC和6mA两大修饰类型,而5mC研究的技术类型有WGBS、RRBS、oxBS、meDIP-seq和hmeDIP-seq,6mA研究的技术类型有6mA-IP-seq。RNA甲基化研究也是有5mc和6mA,使用的技术也是meDIP和meRIP。
不同的甲基化研究技术,使用的不同的技术原理。WGBS主要是通过使用亚硫酸盐处理后,非甲基化的C碱基被转化成U,之后在PCR扩增中被转化成T。这种方法是最经典的全基因组甲基化研究方法,也是甲基化研究的金标准,能得到全基因组范围内的甲基化修饰情况。RRBS主要是利用甲基化敏感性酶来对基因组DNA进行酶切,捕获的区域主要是CpG岛。OxBS技术主要是研究羟甲基化修饰。在实验过程中,需要构建两个文库,一个是常规的WGBS,另一个文库先通过酶来转化甲基化修饰状态的中间态,即通过酶氧化将5hmC氧化成5fc,之后亚硫酸盐处理时,这些甲基化中间状态都能被转化成U,然后通过生信的方法来进行换算。另外一大类就是使用特异性抗体来进行靶向捕获,比如meDIP、hmeDIP和6mA-IP-seq以及meRIP。
3.2 组蛋白修饰研究
组蛋白修饰的研究,主要是研究各种不同的组蛋白修饰参与的染色质调控和基因的表达调控。研究的技术是以ChIP-seq为代表的DNA与蛋白互作的技术。这些技术类型有ChIP-seq、CUT&RUN和CUT&tag。
3.3 染色质重塑研究
染色质重塑研究,主要是研究染色质重塑因子的染色质调控和基因表达调控。研究的技术类型与组蛋白修饰一致,也是ChIP-seq、CUT&RUN和CUT&tag。
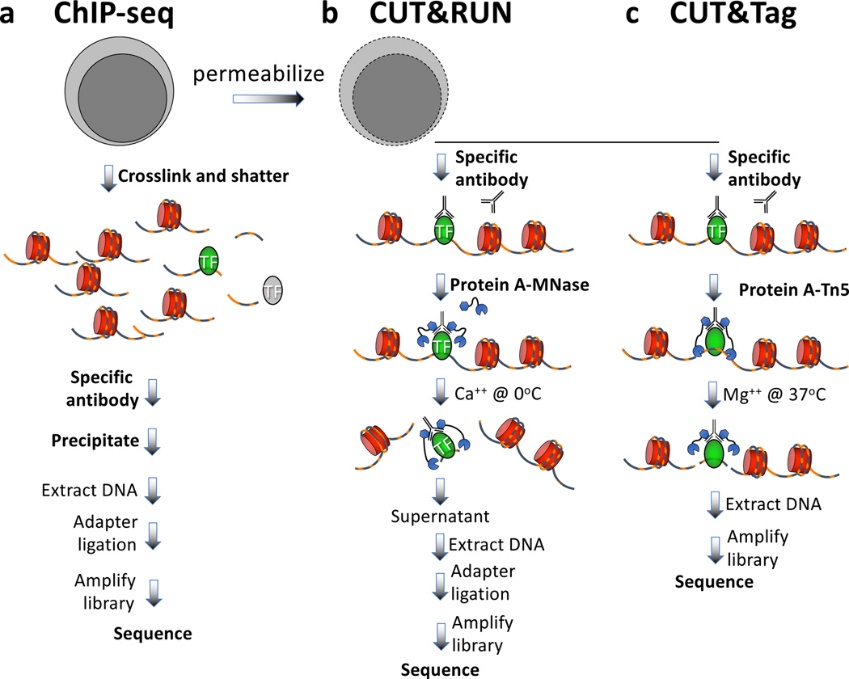
图:DNA-蛋白互作技术流程
除了以上介绍的这些DNA-蛋白互作技术外,还有DAP-seq,也是研究DNA-蛋白互作,但是属于体外实验,适合一些非模式物种的DNA-蛋白互作研究,该实验的优点就是无需抗体,也无需转基因体系,但是因为是体外实验,容易产生很多假阳性。
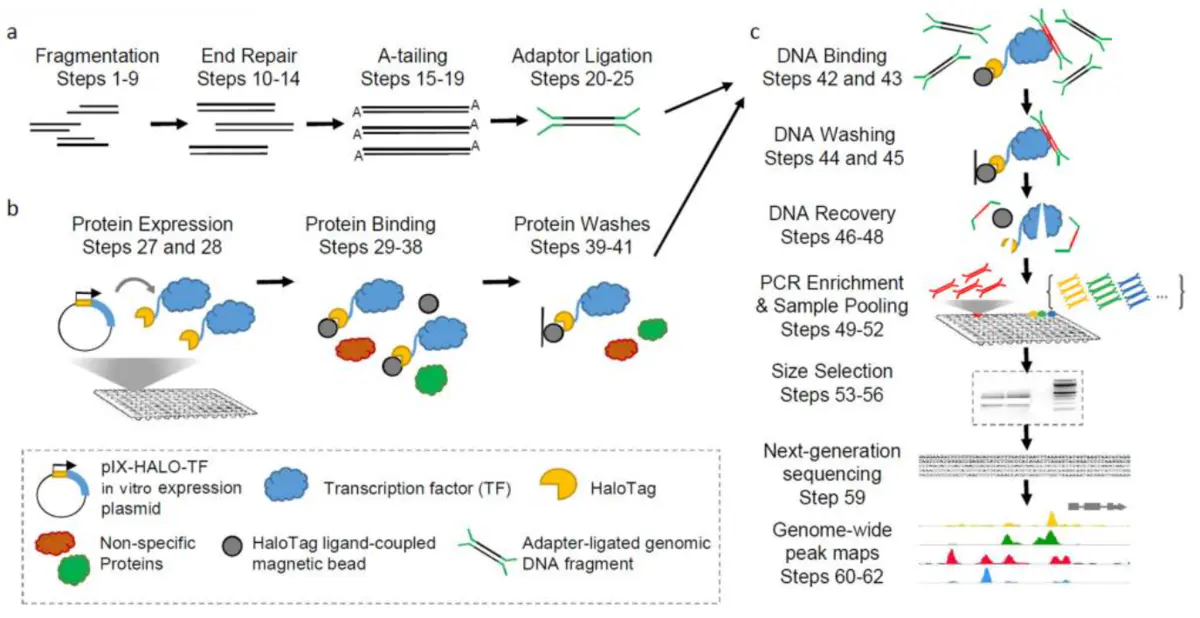
图:DAP-seq实验流程[2]
还有染色质开放性研究技术ATAC-seq,它主要是利用tn5酶来切割染色质开放区域,之后对这些开放区域DNA进行建库后分析。
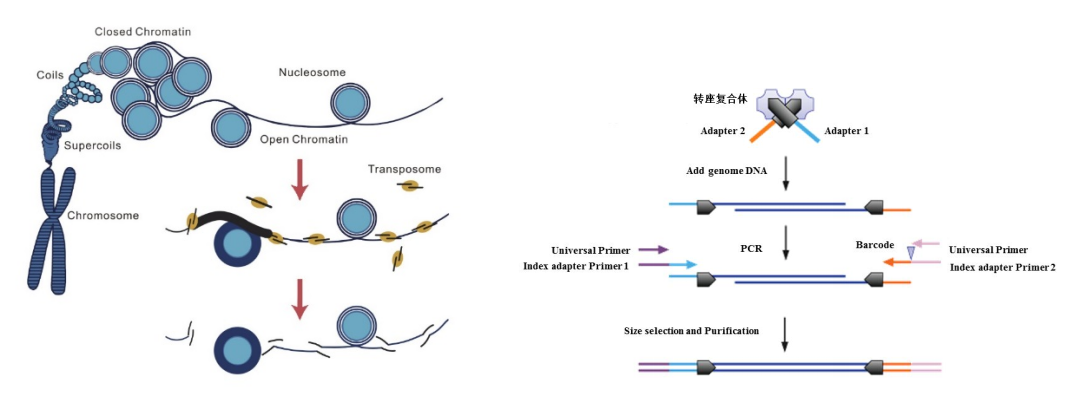
图:ATAC-seq建库流程[3]
3.4 非编码RNA调控
非编码RNA调控,除了有常规的RNA建库外,还有RNA-蛋白互作技术研究。最经典的研究就是关于RNA结合蛋白(RBPs)的研究。主要是通过RNA pull down及RIP-seq/CLIP/miCLIP/eCLIP等技术来实现研究目的。
RNA pull down主要是通过用已知的RNA来pull 未知的互作蛋白,对拉取下来的蛋白进行质谱鉴定。而RIP/CLIP/miCLIP/eCLIP主要是用已知的RNA结合蛋白来调取互作的RNA。通常,我们可以选择RNA pull down和RIP/CLIP/miCLIP/eCLIP同时进行,对需要验证的RNA和RBP进行正向和反向验证。
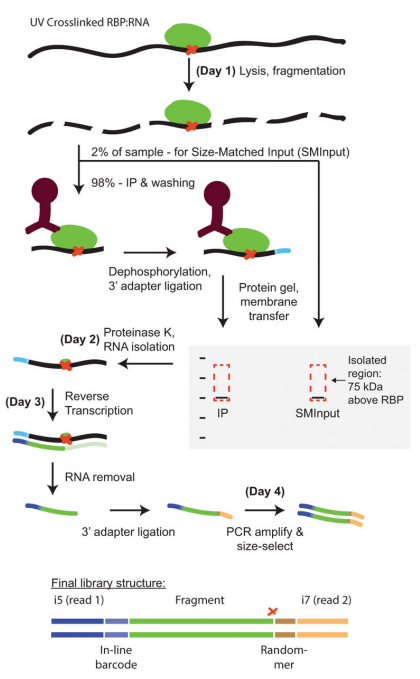
图:eCLIP实验流程【4】
|
产品定位 |
产品名称 |
产品编号 |
规格 |
|
先转化后建库试剂盒 |
12211ES24/96 |
24/96 T |
|
|
机械法建库 |
12199ES24/96 |
24/96 T |
|
|
酶切法建库 |
12204ES24/96 |
24/96 T |
|
|
适用2000以下细胞起始量的ATAC文库构建 |
Hieff NGS® Fast Tagment DNA Library Prep Kit for Illumina® (for 1 ng) |
12206ES24/96 |
24/96 T |
|
适用104-105细胞起始量的ATAC文库构建 |
Hieff NGS® Fast Tagment DNA Library Prep Kit for Illumina® (for 50 ng) |
12207ES24/96 |
24/96 T |
|
CUT&Tag建库试剂盒 |
Hieff NGS® G-Type In-Situ DNA Binding Profiling Library Prep Kit for Illumina® |
12598ES04/12/48 |
4 T/12 T/48 T |
其他模块
|
产品定位 |
产品名称 |
产品编号 |
规格 |
|
短接头-双端index |
Hieff NGS® 384 Dual Index Primer Kit for Illumina® ,Set 1/Set 2(96 种) |
12412ES02 |
96×2 T |
|
长接头-单端index |
13519ES04/16 13520ES04/16 |
48×4 T/16 T |
|
|
UDI接头-双端index |
12404ES01 12405ES01 |
12×2 T |
|
|
单端barcode长接头-MGI |
13360ES02/04/96 13361ES02/04/96 13362ES02/04/96 |
8×2/4 /100 T |
|
|
短接头+UMI UDB primer-MGI |
13367ES02/04 13368ES02/04 |
48×2 T/4 T |
|
|
短接头+UDB primer-MGI |
13451ES02/04 13452ES02/04 13453ES02/04 13454ES02/04 |
48×2 T/4 T |
|
|
DNA纯化 |
12601ES |
/ |
|
|
DNA连接酶 |
10298ES40/42 |
40/400 KU |
|
|
长接头-单端index |
13519ES04/16 13520ES04/16 |
48×4 /16 T |
|
|
接头-双端index |
12414ES02 |
96×2 T |
|
|
短接头+CDI primer-RNA-MGI |
13365ES02 13366ES02 |
96×2 T |
|
|
接头 |
12610ES |
96 index |
|
|
含DNA片段化、末端修复及加A步骤所有模块 |
Hieff NGS® OnePot Pro DNA Fragmentation Reagent 快速片段化/末端修复/A尾添加模块 |
12619ES24/96 |
24 T/96 T |
|
片段化后的DNA末端修复及加A步骤所有模块 |
Hieff NGS® Fast-Pace End Repair/dA-Tailing Module 快速末端修复/A尾添加模块 |
12608ES24/96 |
24 T/96 T |
|
DNA连接酶 |
10298ES40/42 |
40/400 KU |
|
|
核糖体RNA去除试剂盒(人/小鼠/大鼠) |
12253ES24/96 |
24 T/96 T |
|
|
rRNA去除试剂盒(植物) |
12254ES24/96 |
24 T/96 T |
|
|
核糖体RNA去除试剂盒(Human rRNA & ITS/ETS) |
12257ES24/96 |
24 T/96 T |
|
|
人珠蛋白去除探针 |
12806ES24/96 |
24/96 T |
|
|
一步法rRNA去除(人) |
12258ES24/96 |
24/96 T |
|
|
一步法rRNA去除(人) |
12259ES24/96 |
24/96 T |
|
|
一步法人珠蛋白去除(人) |
12260ES24/96 |
24/96 T |
|
|
高效保真的耐U碱基的DNA扩增酶 |
10145ES60/76 |
100 /500 U |
|
|
文库扩增酶模块 |
12620ES24/96/97 |
24/96/400 T |
|
|
2×Super Canace® Ⅱ High-Fidelity Mix for Library Amplification |
12621ES24/96 |
24 T/96 T |
|
|
12624ES24/96 |
24 T/96 T |
参考文献:
[1] Picelli S , Faridani O R , Bjrklund S K , et al. Full-length RNA-seq from single cells using Smart-seq2[J]. Nature Protocols, 2014, 9(1):171-181.
[2]Ecker, Joseph, R, et al. Mapping genome-wide transcription-factor binding sites using DAP-seq[J]. Nature Protocols Erecipes for Researchers, 2017.
[3] Buenrostro J D , Wu B , Chang H Y , et al. ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide[J]. Current Protocols in Molecular Biology, 2015, 109(1).
[4] Nostrand E L V , Pratt G A , Shishkin A A , et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP)[J]. Nature Methods.
