NGS文库构建-关键酶竟然是这些?
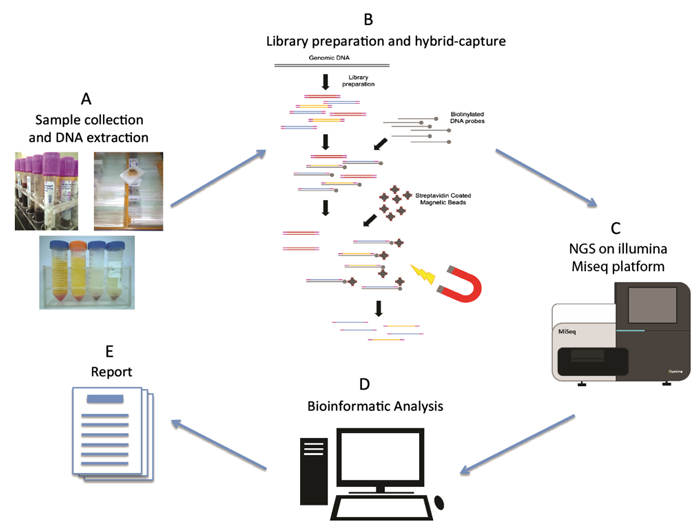
高通量测序又称下一代测序技术(Next-generation Sequencing, NGS),相对于第一代DNA测序技术(Sanger法),它可以同时对几十万乃至数百万条核酸分子序列进行测定,具有通量高、成本低、规模大等显著优势,应用范围非常广泛,目前已经成为全球主流测序技术。
“
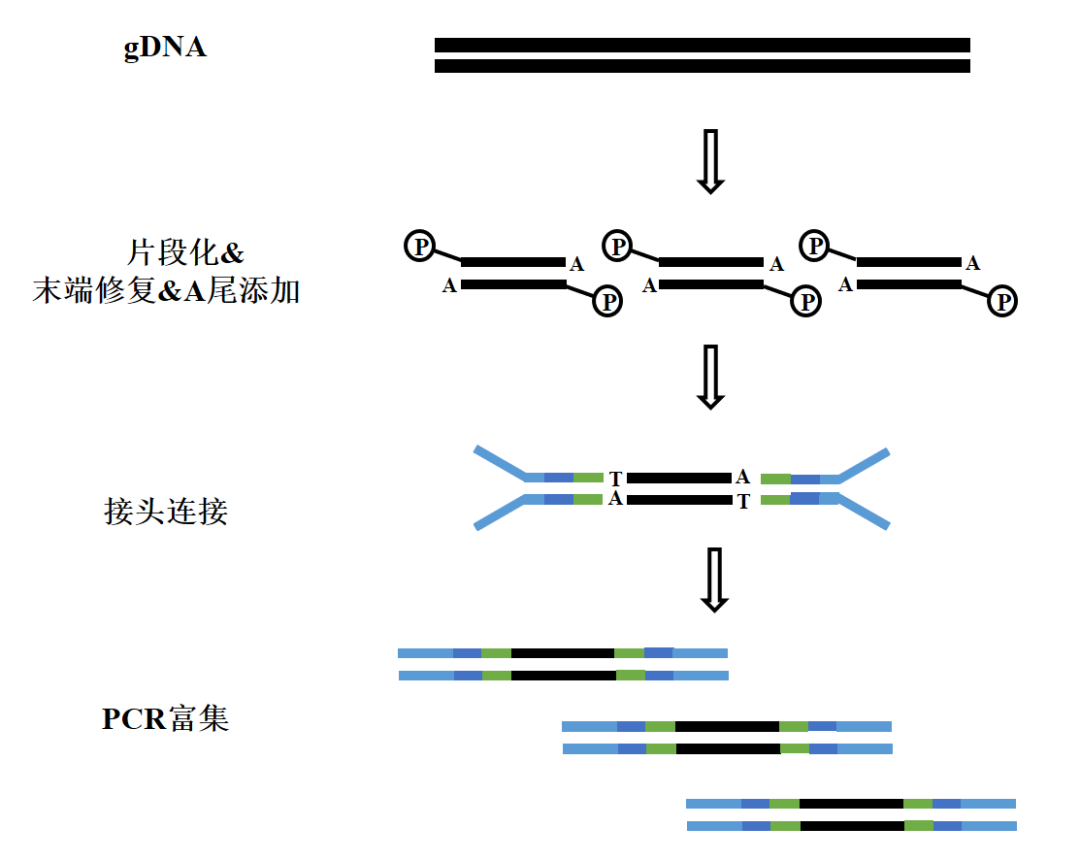
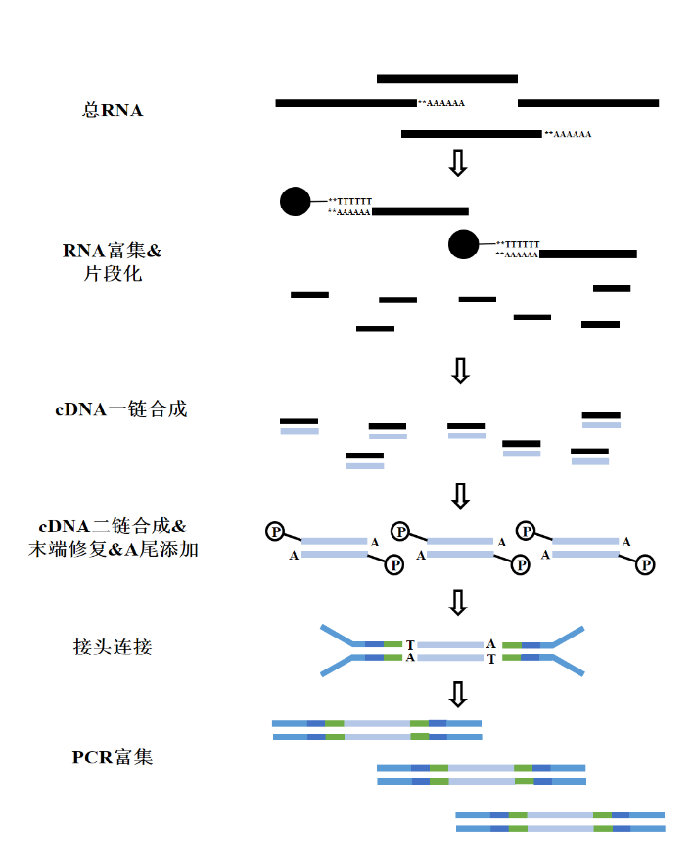
DNA片段化
“
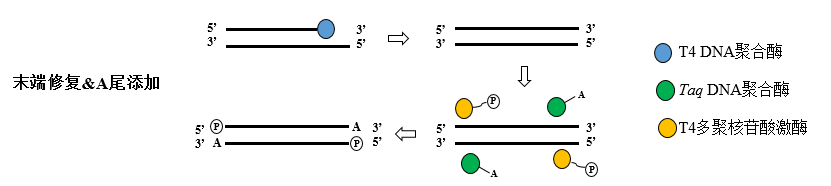
末端修复,3'端加“A”:
“
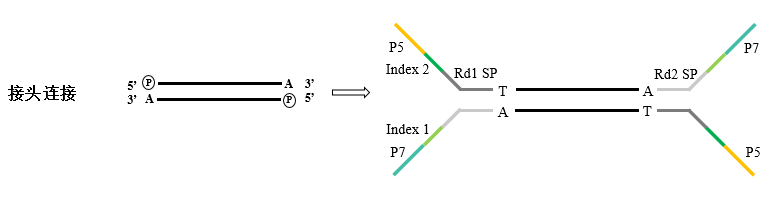
接头连接:
“
PCR富集:
“
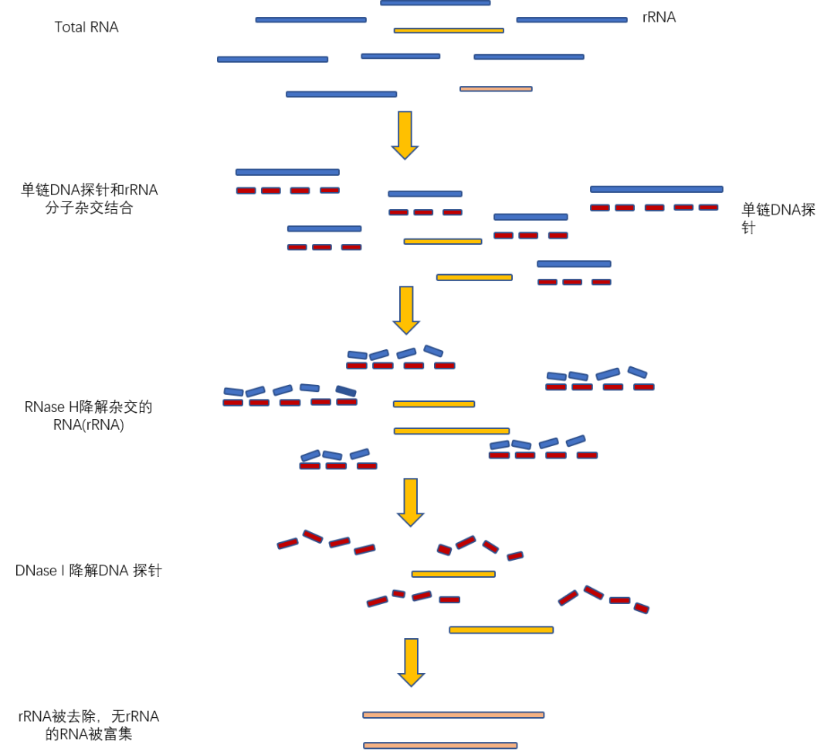
RNA富集:
首先,合成能够与rRNA结合的特异性寡核苷酸探针;
其次,使用能够降解RNA-DNA杂合链中RNA 的RNase H(Yeasen Cat#12906)去除和探针结合的rRNA;
最后,使用可以消化单链或双链的DNA 的DNase I(Yeasen Cat#10325)消化掉DNA探针,从而最终达到去除rRNA的目的。
“
RNA片段化:
“
cDNA一链合成:
“
cDNA二链合成:
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
||
|
|
|
|
|
|
|
|
|
|
|
|
|
||
|
|
Taq DNA Polymerase |
|
|
|
|
|
|
|
|
|
Hieff Canace® Pro High-Fidelity DNA Polymerase |
|
|
|
|
|
Hieff® Smearase Pro |
|
1. Mardis, Elaine R. Next-Generation Sequencing Platforms[J]. Annual Review of Analytical Chemistry, 2013, 6(1):287-303.
2. Gulilat M, Lamb T, Teft W A, et al. Targeted next generation sequencing as a tool for recision medicine[J]. BMC Medical Genomics, 2019, 12.
3. Lundberg K S, Dan D S, Adams M, et al. High-fidelity amplification using a thermostable DNA polymerase isolated from Pyrococcus furiosus.[J]. Gene, 1991, 108(1):1.
4. Miyazaki K. Random DNA fragmentation with endonuclease V: application to DNA shuffling[J]. Nucleic Acids Research, 2002, 30(24):e139.
5. 袁婺洲.《基因工程(第二版)》:化学工业出版社,2019
