干货|你对DNase I“全能选手”了解多少?
“
RNA提取或反转录前去除gDNA
RNA作为实验室常研究的样本,其质量的高低很大程度上会直接影响实验数据的质量。一般在RNA提取过程中无法完全避免gDNA残留,因此,在进行具有挑战性的下游应用(例如mRNA表达量分析,转录组分析等)之前,通常建议对RNA样本进行DNase I处理,消化残留的gDNA。消化gDNA步骤可以在RNA提取期间、RNA提取后或者RNA反转录之前进行。
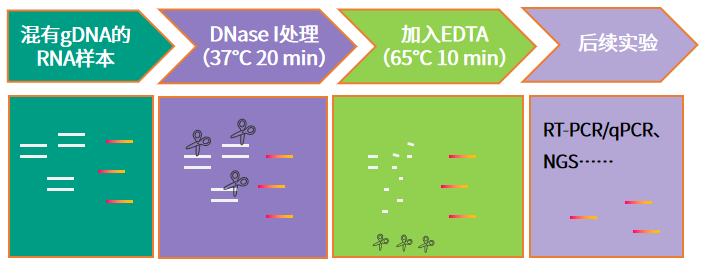
图2:基于DNase I的gDNA去除流程
“
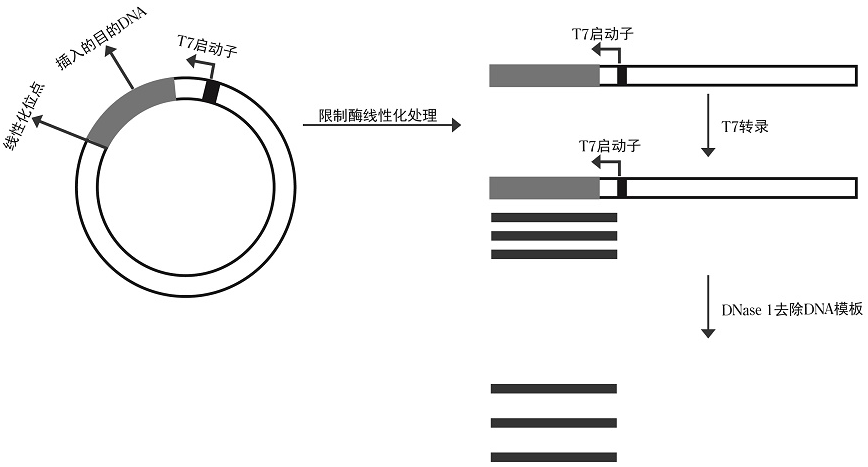
体外转录去除模板DNA
体外转录(IVT)主要是以DNA为模板,在适当的底物和缓冲液的作用下,通过体外转录得到RNA。这一过程中,常用的RNA聚合酶包括T7,T3,SP6等,它们负责催化RNA的合成。然而,合成的RNA产品可能会含有DNA残留,消除DNA残留有利于下游实验的开展。
特别是在mRNA疫苗的研发过程中,去除这些DNA残留是一个非常关键的步骤,它直接关系到后续纯化过程的难易程度及最终产品的纯度。为了高效地消除模板DNA,通常采用DNase I进行处理,以确保RNA样品中不含有DNA残留,这一步骤有助于提高实验的整体准确性和效率。
“
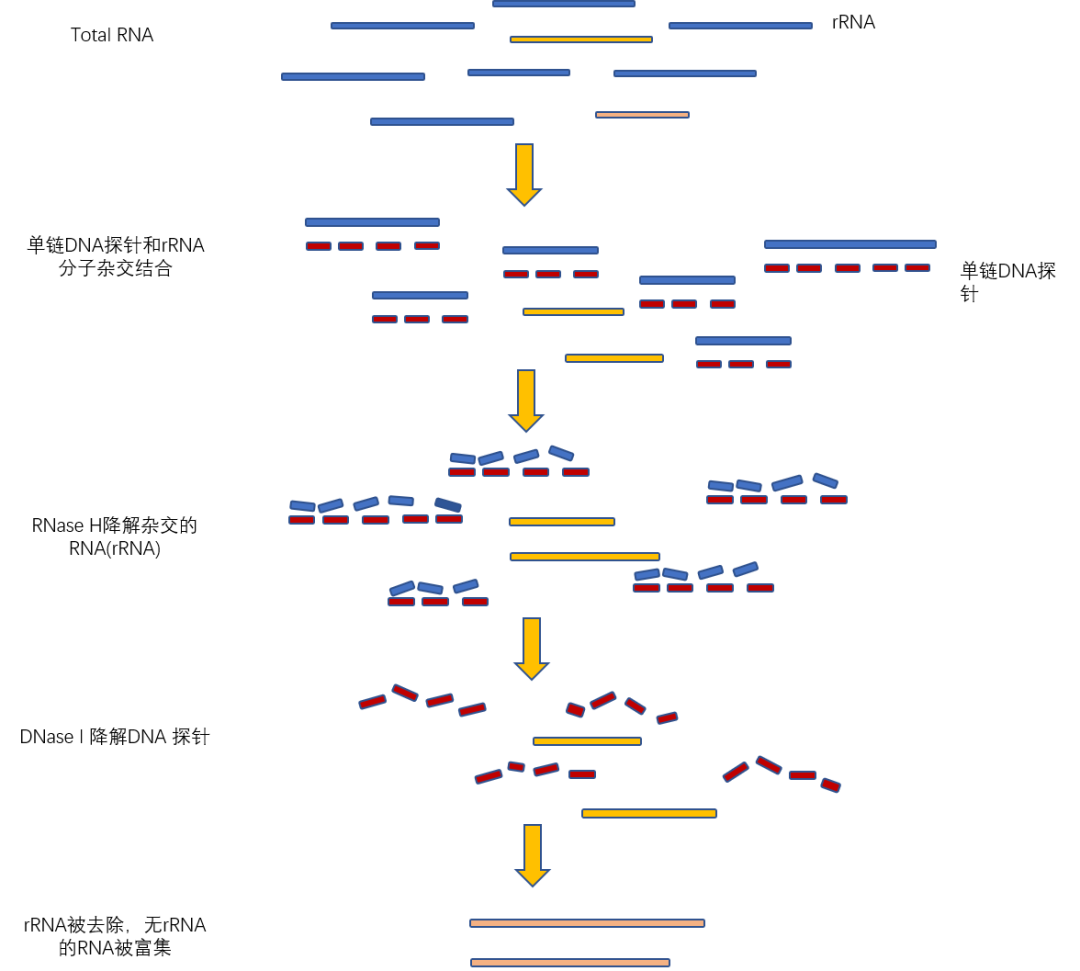
rRNA去除:常应用于RNA建库测序
操作步骤
1.提取Total RNA;
2.单链DNA探针和rRNA分子杂交结合(注:需要设计与合成rRNA 特异性单链 DNA 探针);
3.RNase H降解杂交的rRNA;
4.DNase I降解DNA 探针;
5.rRNA被成功去除,剩下非rRNA的RNA模板。
“
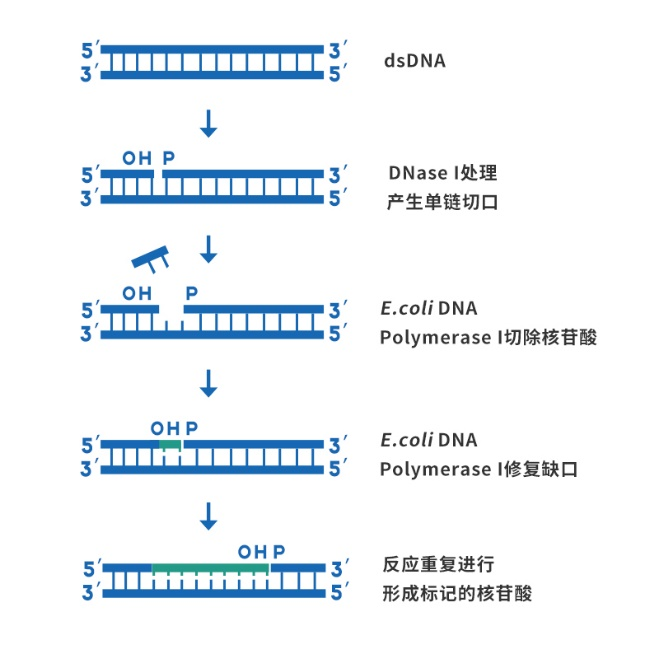
缺口平移法进行DNA标记
主要原理如下:
1.先用适宜浓度的DNase I在待标记的双链DNA的每一条链上产生若干个单链缺口,缺口处形成3’羟基末端。
2.再用E.coli DNA Polymerase I的5’→3’外切酶活性从缺口处5’端切除一个核苷酸,同时E.coli DNA Polymerase I的5’→3’聚合酶活性将一个带标记的核苷酸引入到缺口的3'端,以修补缺口,随着缺口在DNA链上的移动,标记的核苷酸就掺入到新合成的DNA链中。
“
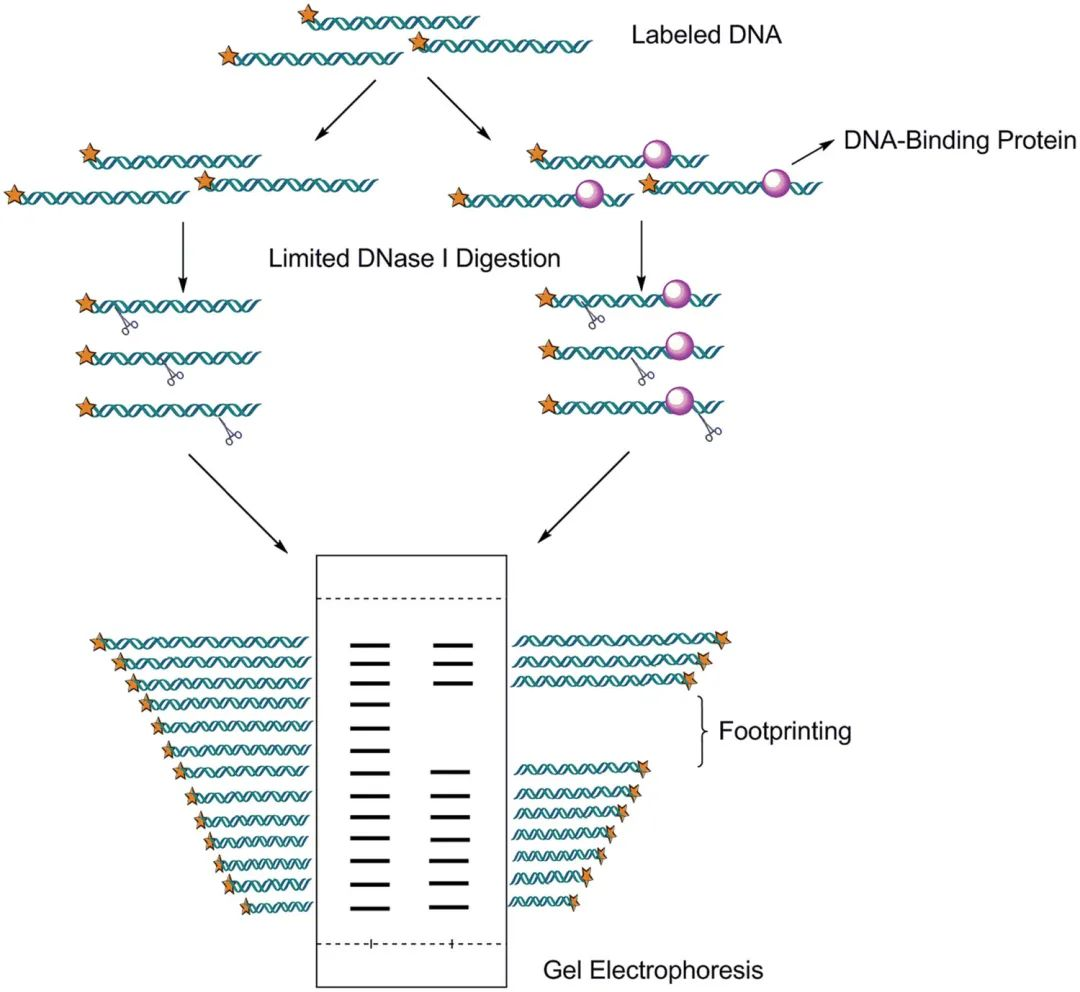
DNase I 足迹法分析实验
实验大致步骤如下:
1.将待检双链DNA分子进行单链末端标记。
2.将蛋白质和DNA混合后,加入适量的DNase I进行酶切,形成不同长度的DNA片段。该过程需要控制酶的用量,最好保证相邻的DNA片段只相差一个核苷酸,并列设置未加蛋白质的对照。
3.从DNA上除去蛋白质,将变性的DNA用PAGE电泳分离,放射自显影,与对照组相比后解读出足迹部位的核苷酸序列。
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
- Ndiaye C, Mena M, Alemany L, et al. HPV DNA, E6/E7 mRNA, and p16INK4a detection in head and neck cancers: a systematic review and meta-analysis[J]. The Lancet Oncology, 2014, 15(12): 1319-1331.
- Broccolo F, Fusetti L, Rosini S, et al. Comparison of oncogenic HPV type‐specific viral DNA load and E6/E7 mRNA detection in cervical samples: results from a multicenter study[J]. Journal of medical virology, 2013, 85(3): 472-482.
- Huang Z, Fasco M J, Kaminsky L S. Optimization of Dnase I removal of contaminating DNA from RNA for use in quantitative RNA-PCR[J]. Biotechniques, 1996, 20(6): 1012-1020.
- Kang D D, Li H, Dong Y. Advancements of in vitro transcribed mRNA (IVT mRNA) to enable translation into the clinics[J]. Advanced Drug Delivery Reviews, 2023, 199: 114961.
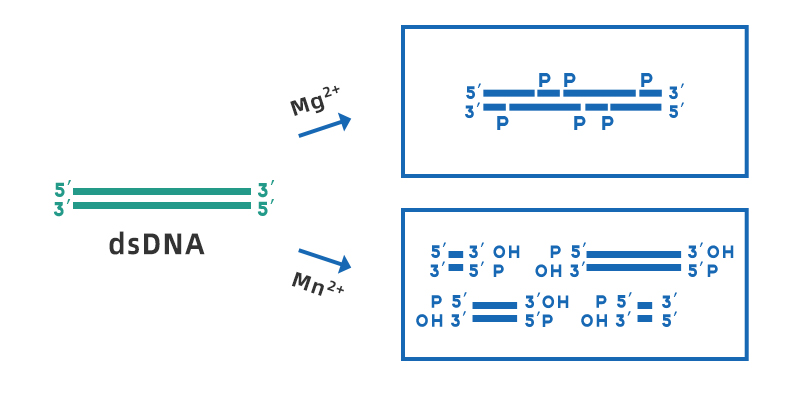
- Wiame I, Remy S, Swennen R, et al. Irreversible heat inactivation of DNase I without RNA degradation[J]. Biotechniques, 2000, 29(2): 252-256.
- Adolph S, Hameister H. In situ nick translation of metaphase chromosomes with biotin-labeled d-UTP[J]. Human genetics, 1985, 69: 117-121.
- Song C, Zhang S, Huang H. Choosing a suitable method for the identification of replication origins in microbial genomes. Front Microbiol, 2015, 6: 1049.
