干货分享|酶切法DNA建库“十问实答”,助您建库无忧
第二代测序(NGS)技术的迅猛发展,使得越来越多的科研工作者将其作为课题研究的主要手段。在NGS过程中,基因文库的质量直接影响后续的测序结果,因此,构建高质量的基因文库是整个测序过程中的关键步骤。酶切法构建DNA文库近年来受到广大科研人员的青睐。然而,在使用酶切法的过程中,大家或多或少会遇到各种问题。接下来,小翌将带领大家一同解决实验开展前后可能遇到的困惑。
实验开展前,“千头万绪”
1、DNA文库构建方法的选择:为什么酶切法建库如此备受青睐?
酶切法构建DNA文库,相较于传统的超声法和转座酶法具有以下优势:
无偏好片段化酶:具有稳定的片段化效果,无需复杂的机械片段化过程。酶切产物片段大小同样本类型、Input DNA量和GC含量等均无相关性,仅与酶切时间有关。
DNA损伤风险低:机械力会引起DNA的物理损伤,尤其是对于脆弱或较少量的样本。酶切法使用的片段化酶是针对特定序列进行切割的,这种生物化学反应通常在较温和的条件下进行,能够最大限度地减少对DNA的物理损伤。
建库流程精简:一步完成片段化/末端修复/加A ,片段化/末端修复/加A (30min)-接头连接(15min)-纯化/分选(30min)-文库扩增(15min)-纯化分选(30min),常规建库预计耗时约2h,PCR-free建库预计耗时1h。
宽泛的样本投入范围:相比较固定投入量,固定酶切时间的TN5建库,酶切法建库适用500 pg-1 μg,满足个性化建库。
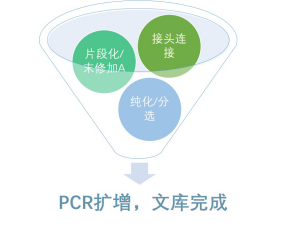
翌圣Onepot系列酶切法建库流程
2、酶切法建库运用的是什么酶,类似限制性酶切酶吗,具体如何打断DNA呢?
不同于限制性酶切酶,片段化酶是随机内切酶,不受特定酶切位点的限制,随机切割对应模板DNA,片段化的产物属于粘性末端,后续需要进行末端修复。
3、酶切法建库适合哪些应用研究,对样本具有普适性吗?
酶切法建库可适用全基因组测序(含PCR-free文库构建)、全基因组重测序、全外显子或其他靶向捕获测序、宏基因组测序等。适合样本类型包含gDNA、cDNA和FFPE样本等,不适合cfDNA(cfDNA无需打断)。
4、酶切法建库试剂盒针对哪些样本建库效果比较好?
兼容范围为1 ng – 1μg Input DNA。应尽可能使用A260/A280= 1.8-2.0的高质量Input DNA。基因组DNA和cDNA均可获得高产高质的文库。
举例:展示为翌圣onepot pro系列12972ES应用于不同物种的文库均一性测试
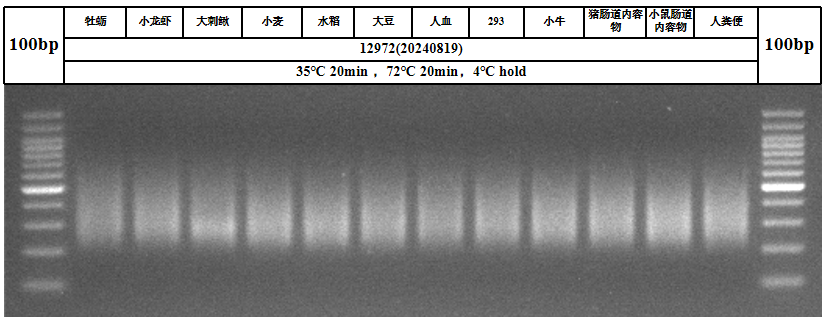
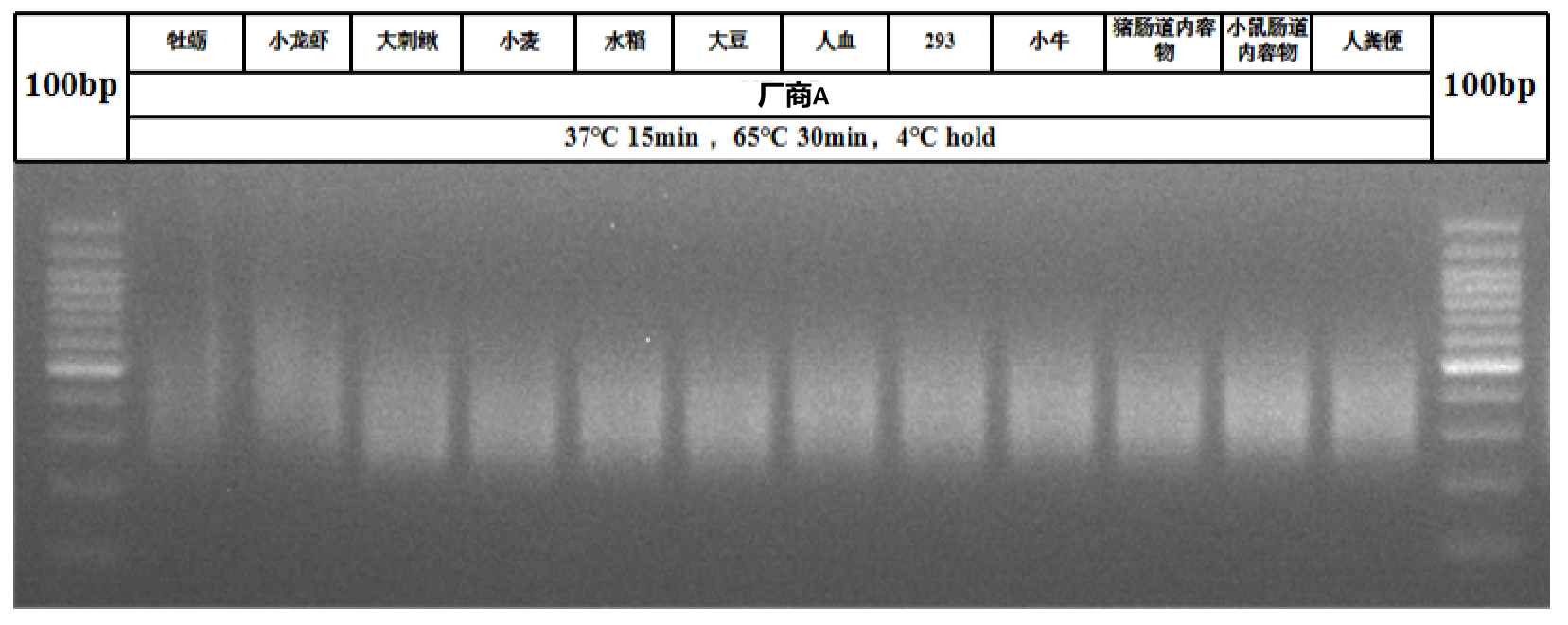
结论:以200 ng不同物种gDNA为模板,使用12972ES和厂商A建库试剂进行文库构建,胶图显示,12972ES在文库均一性上表现优于厂商A。
5、对于不同类型的DNA样本,酶切时间如何控制?
对于常规的高质量基因组DNA,酶切时间如下:
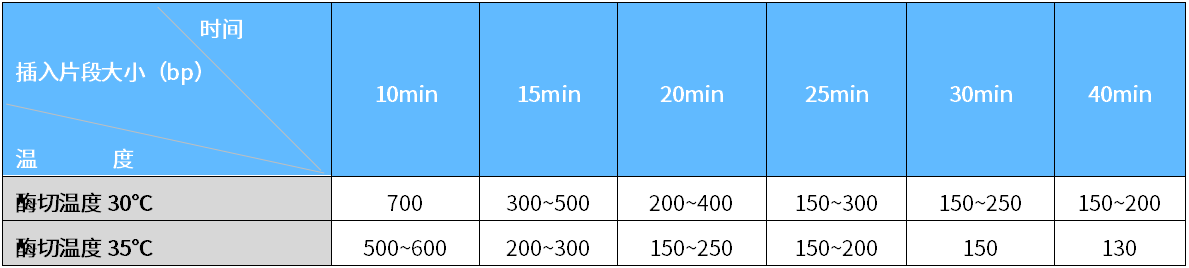
为保证优质精确的片段化效果,建议在自身实验体系中进行微调。
实验开展后“五味杂陈”
6、gDNA片段化参考说明书酶切条件酶切20 min,竟然切不动?
一般优先考虑DNA的质量问题,比如 Input DNA中引入高浓度金属离子螯合剂或其他盐离子,可能会影响酶切效果,建议将DNA稀释在ddH2O或不含EDTA的10 mM Tris Buffer (pH 7.5-8.0)中再进行片段化。其次,如果样本是反转录得到的全长cDNA,则需考虑样本纯度问题。另外,对于环状DNA的酶切,需要另行摸索酶切时间体系。特殊样本可考虑磁珠纯化之后进行酶切或适当延长酶切时间。
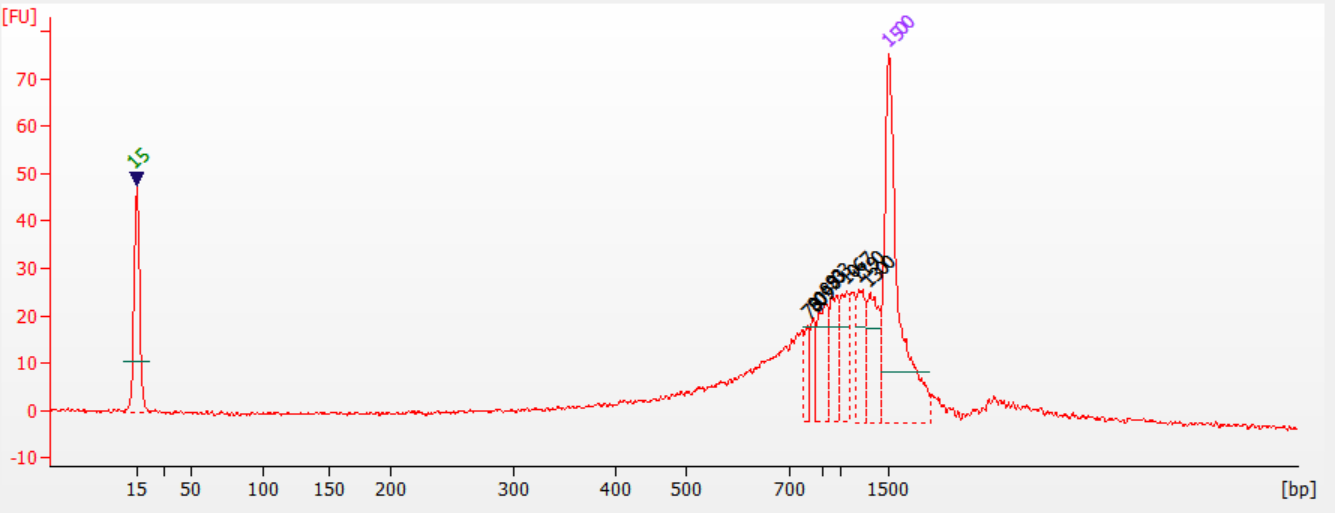
7、FFPE样本,酶切时间太难控制了?
众所周知,FFPE样本虽然使组织得以长期保存,但其特殊的制作方法和保存过程对核酸分子造成多方面的影响:核酸与蛋白交联、降解严重等诸多问题。我们先来看下不同质量的FFPE样本,采用相同的酶切条件影响有多大。
举例:相同酶切条件下,质量高的样本酶切效果较好,满足需求(样本4),质量差的样本出现过度酶切现象,酶切片段偏小(样本1/3)。
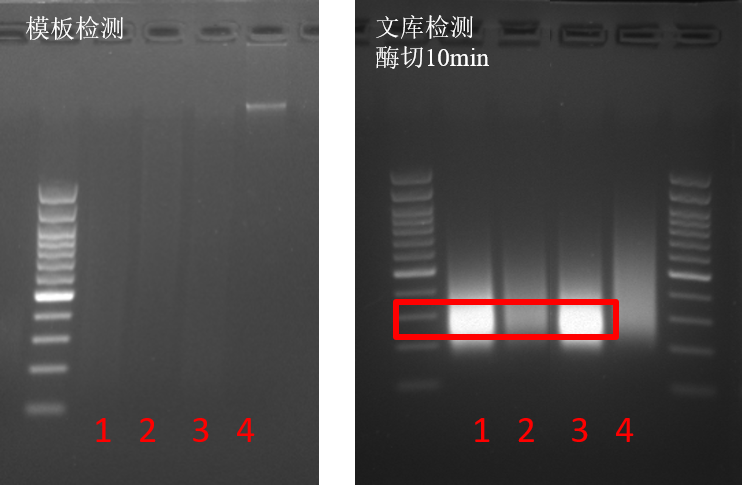
因此对于不同降解程度的FFPE样本,建议做好质量控制,对样本进行分层级,不同层级的推荐酶切时间参考下表。
|
插入片段主峰大小 |
片段化时间 |
DIN* |
|
250 bp |
9-13 min |
> 8.0 |
|
250 bp |
8-11 min |
6.5-8.0 |
|
250 bp |
4-8 min |
4.2-6.5 |
|
250 bp |
3-6 min |
2.5-4.2 |
备注:FFPE样本DNA质量也可通过琼脂糖凝胶电泳胶图简单判断,高质量样本-DNA质量大于10kb,条带明亮单一,建议酶切10 min左右;一般质量样本-胶图显示无明显主带,整体弥散条带,建议酶切6-8 min;质量较差样本-胶图无主带,整体条带弱,建议酶切2 min;极差FFPE样本,建议可不酶切,搭配FFPE修复试剂直接建库。
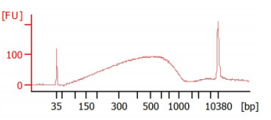
8、DNA酶切后文库出现大片段残留?
酶切文库出现大片段残留现象,考虑Input DNA纯度外,建议适当延长酶切时间或提高酶切温度。另外,也需要选择合适的循环数,循环数过高会导致过度扩增,过度扩增文库会出现大片段残留。
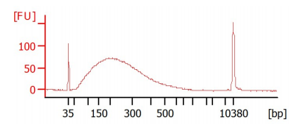
9、酶切时间也不长,竟然过度酶切了?
以插入片段300 bp为例,白细胞gDNA,30 ℃酶切20 min,出现如下图过度酶切现象。建议参考酶切条件需要考虑不同样本gDNA大小,如白细胞gDNA较小,应适当缩短酶切时间,否则易出现如下过度酶切现象。
10、酶切法建库,如何做到较好的质量控制?
1)文库浓度检测:qubit法或qPCR
2)文库长度检测:琼脂糖凝胶电泳、Agilent 2100/2200或Qseq
3)文库质量鉴定:Agilent 2100/2200或Qseq
4)文库大小分布、引物二聚体和过扩增产物的缺失,可通过电泳方法进行确认
构建优质的DNA文库不仅需关注上述因素,还需选择高质量的建库产品。翌圣生物为满足广大科研工作者的NGS建库实验需求,推出了全面的NGS建库解决方案,方便大家根据具体实验需求进行选择。
|
试剂类别 |
产品名称 |
货号 |
规格 |
应用场景 |
|
酶切法 |
Hieff NGS® OnePot Pro DNA Library Prep Kit V4 |
12972ES08/24/96 |
8/24/96 T |
科服、育种 |
|
12194ES08/24/96 |
8/24/96 T |
肿瘤、遗传病 |
||
|
12316ES24/96 |
24/96 T |
病原mNGS |
||
|
机械法 |
Hieff NGS® DNA Library Prep Kit 2.0 |
12927ES08/24/96 |
8/24/96 T |
各场景通用 |
|
磁珠 |
12601ES03/08/56/75 |
1/5/60/450 mL |
DNA纯化分选 |
|
|
接头 |
12412ES02 |
96×2 T |
序列标识 |
|
|
13361ES02/04/96 |
16×2 T/16×4 T/16×100 T |
|||
|
定量 |
12642ES60/76 |
100/500 T |
DNA浓度测定 |
