掌握这几个影响因素,你的反转录实验就完美了
小翌君前几期一直在和大家聊qPCR,今天我们开启一个新的话题,聊一聊反转录。
反转录(reverse transcription)定义:
反转录过程简单来说是以RNA为模板合成DNA的过程,是对中心法则的重要修正和补充。
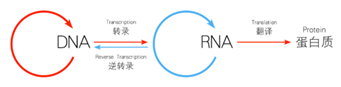
中心法则
通常会出现两个不同的表述,逆转录和反转录,二者的本质区别在于:反转录是在研究过程中人为提取RNA并以之为模板人工合成DNA的过程。逆转录是RNA病毒自主行为,以RNA为模板形成DNA的过程。前者发生在体外,后者发生在体内。反转录反应的示意图如下图所示:
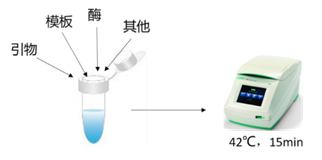
反转录实验过程
看似简单的反转录实验,但也受到多种因素的影响,如模板、引物、酶以及反应条件等。接下来小编将带您对其各个击破。
一、模板
① RNA自身结构(高GC、复杂二级结构)
RNA由于自身序列折叠,具有不同复杂的二级结构:配对的双链RNA呈螺旋,发卡环,突环,内环,多分支环等结构。由于配对碱基不同,其配对的稳性也不同,如G与C之间的配对,三对氢键,最为稳定。A与U为两对氢键相互作用;G与U为一对氢键相互作用,最不稳定。故对于RNA模板来讲,GC含量越高,二级结构越为复杂。
在进行反转录的过程中,如下图所示,当引物延伸到有二级结构的地方,反应就会被迫终止,影响cDNA的合成长度。此时可建议先将模板进行65℃孵育5min然后迅速冰上冷却使二级结构变性;同时可通过在较高的温度下(如55℃)反应以降低RNA二级结构的形成。
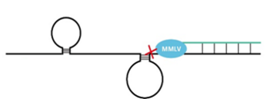
反转录实验延伸过程
② 纯度和浓度的测定
RNA纯度会很大程度地影响反转录实验,如RNA纯化过程中混入的盐、金属离子、乙醇、苯酚等均是常见的逆转录酶抑制剂。此外植物组织中的多糖多酚和腐殖酸等也会对逆转录酶造成抑制作用。
对于RNA纯度的测定,通常会选用Nano drop进行测定。纯度完好的RNA:1.8<OD260/OD280<2.0(<1.8时表明有蛋白质或酚污染,可增加酚抽提;>2.0时表明可能有异硫氰酸残存);OD260/OD230应大于2。(<2表明有异硫氰酸胍,β-巯基乙醇或乙醇的残留;可进行再次沉淀,重复乙醇洗涤)。
对于RNA浓度的测定,通常也会选用Nano drop进行测定。
③ 完整度
RNA的完整度在很大程度上影响着cDNA合成的长度与质量。在进行RNA提取处理储存和实验过程中需要采取特殊的保护措施,如操作者佩戴手套和口罩,全程使用无RNA酶污染的实验耗材等。
对于下游的克隆实验,RNA完整度将会影响cDNA的合成长度,从而影响目的基因的合成。对于下游的目的基因定量实验,将会严重影响其CT值,从而影响定量结果精准度(见下图)。
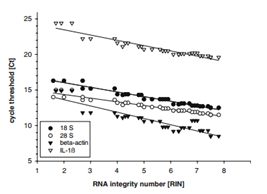
RNA RIN值和CT值的关系
RNA的完整度通常会通过琼脂糖凝胶电泳进行检测,对于28S和18S rRNA的比值来评估RNA的完整度,接近2:1为较为完整的RNA。
目前,Agilent 2100生物分析仪已逐渐成为RNA样品质量控制(QC)的行业标准,在分析RNA完整性之后给出准确的数值(RIN值),该值在8-10之间表示RNA质量非常好,小于7时表示RNA完整度较差。
④ gDNA的去除
在进行下游qPCR实验过程中Totol RNA中混入的gDNA可直接作为PCR反应的模板进行扩增,造成实验结果的假阳性。因此在反转录之前必须要对RNA模板进行去gDNA处理,保证模板中仅有cDNA。
那么如何解决呢?
我们可通过加入DNaseⅠ去除gDNA。但是在反转录实验前需将DNaseⅠ灭活(通过高温加热或加入EDTA),否则该酶会将cDNA降解,但是高温灭活DNaseⅠ会造成RNA的降解和损失,加入EDTA灭活又会引入新的RNA酶抑制剂。
Yeasen生物提供的反转录试剂盒含gDNA digester组分,在反转录之前可将其与RNA模板混匀42℃,2min即可去除gDNA组分,同时此试剂盒中的SuperMix 可终止gDNA digester的作用,保证cDNA的完整性。
二、引物
① Oligo(dT)
Oligo(dT)引物是约12-18个多聚胸腺嘧啶T组成的重复寡核苷酸序列,能够特异性地与真核生物mRNA的ploy(A)尾退火,故不适合缺少ploy(A)尾结构的RNA,如原核生物RNA或microRNA,也不适用于已降解的RNA,如FFPE样品中RNA。
真核生物中mRNA仅占总RNA的1%-5%左右,通常在进行从真核生物中构建cDNA文库,或者克隆全长cDNA以及3’RACE等研究时会优先选择 Oligo(dT)引物。
对于复杂模板RNA,如含较多二级结构,在进行cDNA的合成延伸过程中,当延伸到二级结构处时,可能会造成延伸终止,若选择Oligo(dT)可能会造成mRNA 5’端信息的丢失。
② Random N6-N9
随机引物是由随机碱基组成的寡核苷酸,通常由6个核苷酸组成,称为随机6聚体。该种引物不具备特异性,rRNA, tRNA,非编码RNA,小RNA,降解RNA,复杂二级结构的RNA等都可以作为其模板。该类引物可提高cDNA的产量和浓度,但是会使合成的cDNA长度变短。
③ 基因特异性引物
基因特异性引物能够与模板特异性互补,产生目标性很强的cDNA,对于后续的PCR扩增更特异,适用于已知目的序列。通常应用于一步RT-PCR反应。当使用基因特异性引物(GSP)无法有效合成第一链cDNA时,可选用Oligo(dT)或随机引物。
三、酶
不同的逆转录酶具有不同的功能活性,表现出不同的逆转录效率,如合成cDNA的长度,产量,对复杂模板的适应程度等。通常共有的活性如下:
1)RNA指导的DNA聚合酶活性:以RNA为模板,催化dNTP聚合形成DNA的过程。
2)RNaseH活性:在反应过程中切割RNA:cDNA杂合链中的RNA模板。降低此活性,可避免在合成较长cDNA之前模板RNA被降解掉。
3)DNA指导的DNA聚合酶活性:以反转录合成的第一条cDNA单链为模板,再合成第二条cDNA分子。
4)热稳定性:此性能是影响cDNA合成的重要因素。升高反转录过程中的温度,有助于高GC或复杂二级结构RNA模板的变性,实现全长cDNA的合成。
5)保真性:RNA反转录为cDNA过程中的序列精确度。由于逆转录酶不具备3’-5’外切酶活性,因此没有校正功能,所以合成的cDNA出错率较高。通常,除测序对精准度要求较高外,其他情况下,反转录中引入的错误数量可忽略不计,因为cDNA中的错误不会被放大。
6)抗抑制能力:当模板纯度较低、抑制物较多时,抗抑制能力强的逆转录酶才能正常进行cDNA的合成。
目前使用的逆转录酶大都为MMLV(来源于莫洛尼鼠白血病病毒)。未经改造的MMLV最适反应温度为37℃,有较低的RNaseH活性。Yeasen生物对MMLV反转录酶进行改造,提高其最适反应温度(高温下有利于RNA二级结构的打开),降低RNaseH活性,提高cDNA合成长度,增强其灵敏度和耐抑制能力。改造结果见下图:
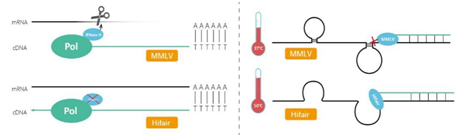
逆转录酶的改造
各种逆转录酶的性能参数如下表:
|
酶类 |
AMV逆转录酶 |
MMLV逆转录酶 |
Yeasen逆转录酶 |
|
RNaseH |
高 |
中 |
低 |
|
反应温度 |
42℃ |
37℃ |
55℃ |
|
反应时间 |
60min |
60min |
20min |
|
cDNA合成长度 |
≤5kb |
≤7kb |
≤12kb |
|
产量 |
中 |
低 |
高 |
反应过程
反转录温度通常推荐使用42℃,对于高GC含量模板或者复杂模板,可将反转录温度提高到50℃。反转录产物可立即用于qPCR反应,也可-20℃短期保存;若需长期保存,建议分装后,于-80℃保存,避免反复冻融。
总之,高效率的逆转录反应建立在高质量的RNA模板、逆转录酶,合适的引物,适宜的反应条件等基础上的。实验过程中要注意到所有的影响因素,才能合成出适用于下游实验的高质量cDNA。
往期精彩回顾
理性阐述qPCR实验的Ct值的合理范围(附Ct值过大或过小的解决方法)
