翌圣镁孚泰突破性成果:低dsRNA T7 RNA聚合酶突变体,助力mRNA疗法研究!
随着生物技术的飞速发展,mRNA疗法作为一种新兴的治疗手段,因其可编程性强、响应速度快等优势备受关注。在mRNA疫苗和基因治疗等领域中,高效合成高质量的mRNA是实现疗法目标的关键步骤。而在这一过程中,T7 RNA聚合酶扮演着至关重要的角色,它能够高效地催化体外合成mRNA的过程。
尽管T7 RNA聚合酶在mRNA合成中发挥着重要作用,但实际操作中经常会产生双链RNA(dsRNA)这一副产物。dsRNA的存在不仅降低了mRNA的产量和纯度,还可能激发非特异性免疫反应,影响疗效和安全性。因此,减少dsRNA的产生成为了行业的一个迫切需求。
目前,降低dsRNA的方法主要包括优化反应条件和使用辅助酶等。这些方法虽然可以在一定程度上减少dsRNA的产生,但往往伴随着操作复杂、成本提高等问题。与之相比,通过直接改造T7 RNA聚合酶来降低dsRNA的产生则是一种更为根本的解决方案。酶定向改造技术可以通过精确改变酶的氨基酸序列或结构,从而改善其催化特性,提高产物的纯度和产率。对于T7 RNA聚合酶而言,针对性的改造不仅可以减少dsRNA的产生,还能提升合成mRNA的整体效率和质量。
作为mRNA体外合成原料供应商(了解更多),翌圣生物凭借子公司镁孚泰生物的ZymeEditor定向进化平台的能力,持续在进化mRNA体外合成的核心酶原料——T7 RNA聚合酶。为了尽可能消除体外转录过程产生的dsRNA等副产物,镁孚泰生物进化团队通过理性设计与定向筛选结合的方式对T7 RNA聚合酶进行改造。
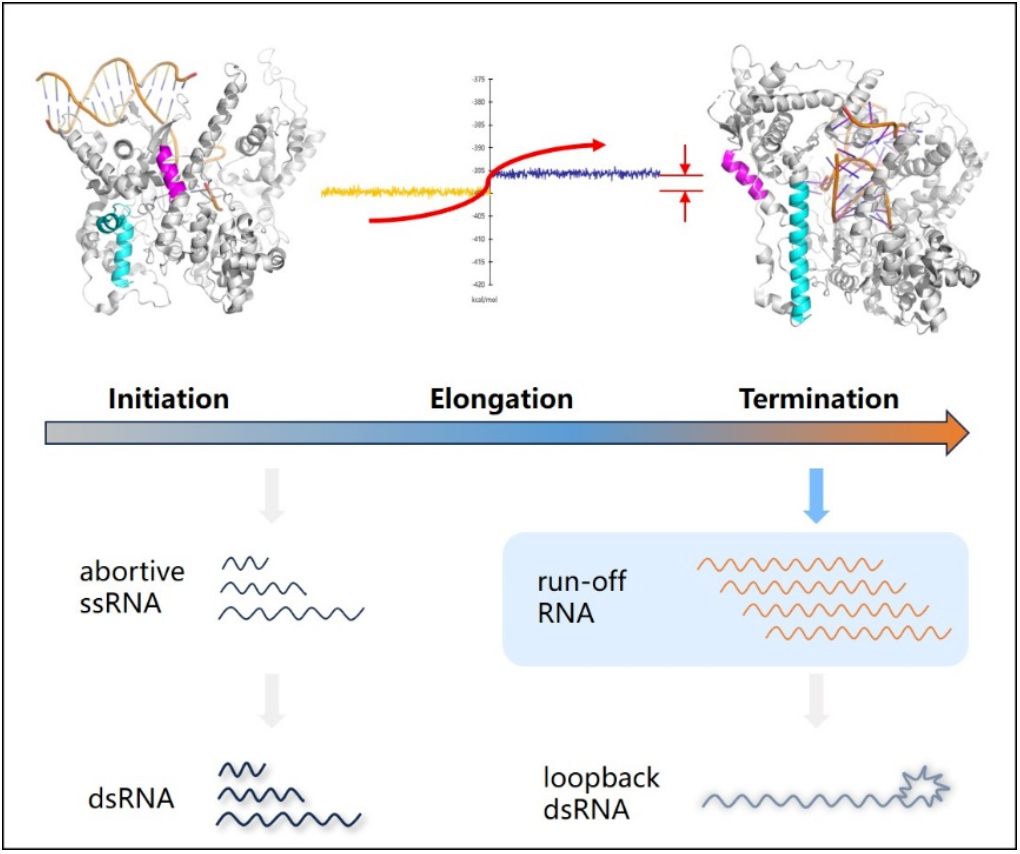
图1. 能垒示意图及dsRNA形成原理
在进行T7 RNA 聚合酶改造过程中,镁孚泰生物一方面通过结构及自由能分析,发现在T7 RNA聚合酶构象变化的同时也伴随着能量的变化,且延伸构象的自由能显著高于起始构象,意味着转录过程需要越过较高的能垒才能产生完整的mRNA链。高能垒不利于构象平稳转变,为此,团队借助虚拟筛选手段锁定了20余个热点氨基酸,并构建了相应的定点饱和文库。另一方面,考虑到关于T7 RNA聚合酶的末端转移及RDRP活性相关的功能、位点及结构域的报导较少,且由于功能相近这两种活性极可能与T7 RNA聚合酶主反应的转录活性(即DNA依赖的RNA聚合活性,DDRP)共享关键位点,难以找到热点氨基酸进行定点改造。因此,镁孚泰生物同时构建了基于易错PCR的数个随机突变文库,结合ZymeEditor平台的进化通量,单个文库的多样性超过106。
为了兼顾T7 RNA聚合酶的产量并监测体外转录反应中dsRNA的含量,镁孚泰生物参照优化后的转录模板,精心设计了多个分子信标(FRET RNA探针),分别靶向目标mRNA产物的不同区域,将反应物产量、完整度以及体系内dsRNA含量等信息与荧光信号关联,体系的荧光强度越强,突变体的性能越有优势。理论上,该筛选方法可按不同探针的荧光强度排序,分别获得高产或Abortive RNA减少的T7 RNA聚合酶突变体,以及完整度提升或loopback dsRNA降低的T7 RNA聚合酶突变体。将反应模板更换为RNA时还可负向筛选突变体的RDRP活性,提升T7 RNA聚合酶的模板选择能力。此外,在液滴反应体系中添加修饰核苷酸、帽类似物,以及提高反应温度还可以进一步筛选耐受非天然底物、热稳定性提升的T7 RNA聚合酶突变体。
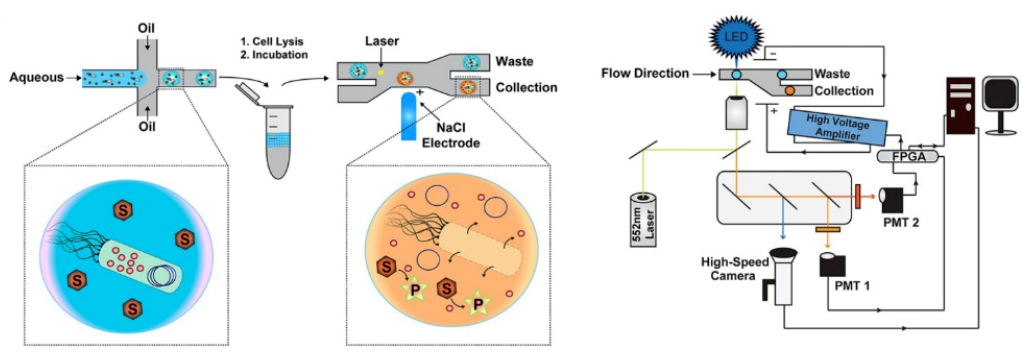
图2. FADS筛选流程【5】
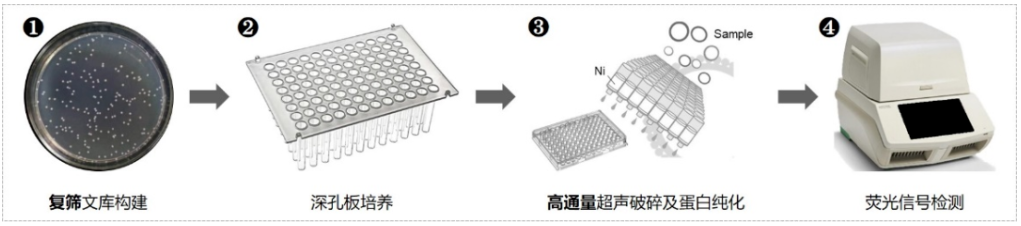
图3. MTPS筛选流程
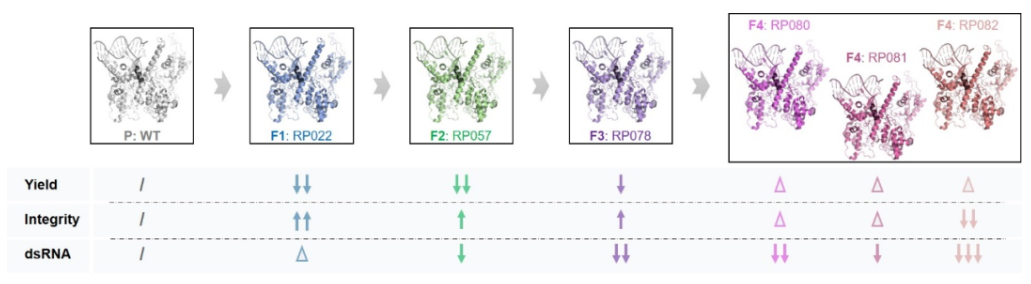
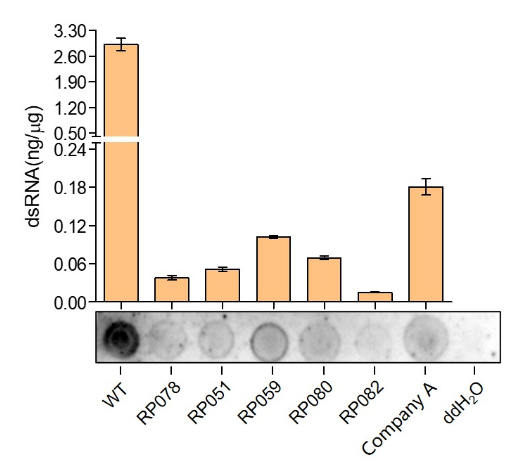
图4. 酶进化历程及突变体性能表征
客户试用满意度高
目前我们还有少量试用名额,如您需要试用或者改造酶的其他性能,点击阅读原文或在线填写需求表!

镁孚泰生物科技(上海)有限公司(镁孚泰生物,Molefuture)是翌圣生物科技(上海)股份有限公司旗下的全资子公司,是一家以酶进化技术为驱动、专注于提供酶改造定制化解决方案的创新型企业。依托于翌圣生物,镁孚泰生物现有六大技术平台:ZymeEditor创新酶进化平台、多宿主高效表达平台、发酵工艺开发平台、纯化工艺开发平台、超洁净酶生产平台以及酶分析与质控平台。基于这六大技术平台,镁孚泰生物可提供酶定向改造、新酶开发、酶工艺开发以及GMP级别规模化生产的全套定制解决方案,以满足酶在体外诊断、生物医药、合成生物、医疗美容、医药中间体等领域的应用需求。
关注镁孚泰官方微信公众号,获取更多酶改造知识!
参考文献:
[1] Sousa R, Mukherjee S. T7 RNA polymerase[J]. Progress in nucleic acid research and molecular biology, 2003: 1-41.
[2] Steitz T A. The structural changes of T7 RNA polymerase from transcription initiation to elongation[J]. Current opinion in structural biology, 2009, 19(6): 683-690.[3] Vallejo D, Nikoomanzar A, Paegel B M, et al. Fluorescence-activated droplet sorting for single-cell directed evolution[J]. ACS synthetic biology, 2019, 8(6): 1430-1440.
[4] Gholamalipour Y, Karunanayake Mudiyanselage A, Martin C T. 3′ end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character—RNA-Seq analyses[J]. Nucleic acids research, 2018, 46(18): 9253-9263.
[5] Vallejo D, Nikoomanzar A, Paegel BM, Chaput JC. Fluorescence-Activated Droplet Sorting for Single-Cell Directed Evolution. ACS Synth Biol. 2019 Jun 21;8(6):1430-1440.
