关于USER酶混合物的应用,看这篇就够了!
尿嘧啶特异性切除试剂(Uracil-Specific Excision Reagent , USER)是由尿嘧啶DNA糖基化酶(Uracil DNA Glycosylase, UDG)和核酸内切酶 Ⅷ(Endonuclease VIII, Endo Ⅷ)(戳链接了解详情)两种酶混合而成。UDG识别ssDNA或dsDNA上的dU碱基并形成AP位点,但磷酸二酯骨架结构保持完整。Endo VIII的裂解酶活性能够使AP位点的3′和5′端的磷酸二酯键断裂,释放无碱基的脱氧核糖,形成单核苷酸间隙。因此,USER酶混合物能在DNA的dU位置产生一个单核苷酸缺口,适用于ssDNA、dsDNA及环状DNA,可应用于
-
RNA链特异性文库构建;
-
尿嘧啶特异性删除介导的克隆(USER-LIC);
-
TALE基本单位的组装;
-
单细胞多个cDNA串联建库。
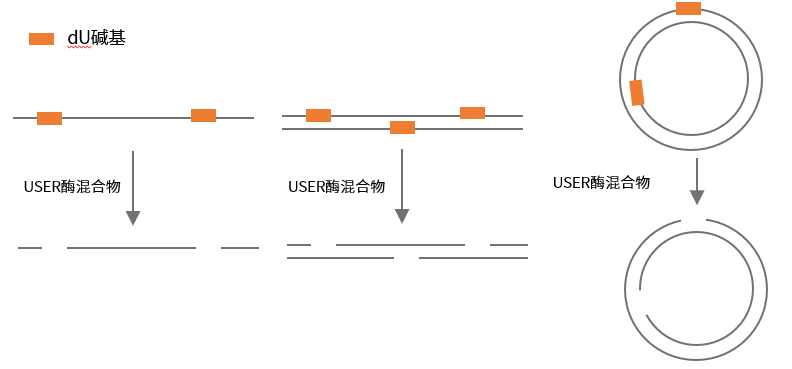
图1. 切除dUTP示意图
Uracil-Specific Excision Reagent的应用
RNA链特异性文库构建
技术路线:
使用引物合成RNA对应的cDNA第一链;
使用dUTP取代dTTP合成cDNA的第二条链;
末端修复、3’加A、接头连接;
去除含dUTP的cDNA链:用USER酶混合物处理,将含有dUTP的cDNA链去除,留下cDNA一条反链;
PCR 扩增,上机测序。
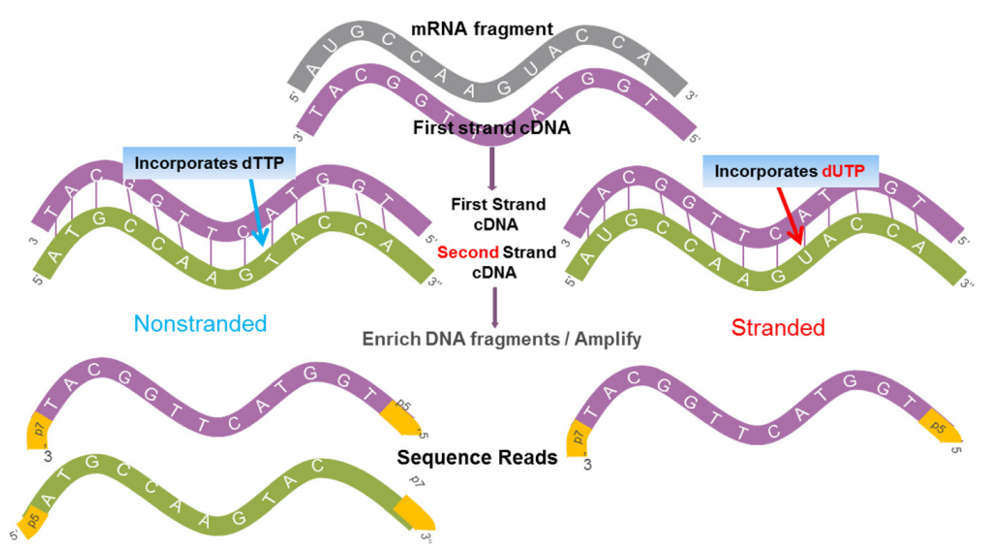
图2. 常规RNA建库与链特异性文库构建对比[1]
尿嘧啶特异性删除介导的克隆(USER-LIC)
技术路线:
扩增:用含dUTP的引物扩增目的片段和载体;
切除dUTP:将得到的两种线性片段用USER酶混合物处理,使它们暴露互补的粘性末端;
连接与转化:将带有互补粘性末端的两种线性片段混合后转化到大肠杆菌感受态细胞中,经过筛选获得目标质粒。
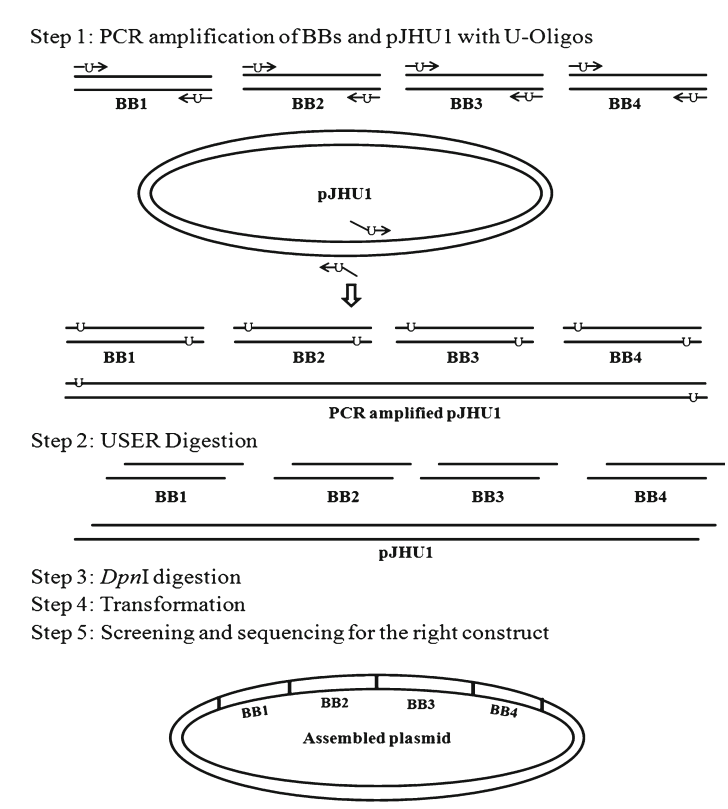
图3. 克隆示意图[2]
助力TALE技术,构建TALE基本单位
技术路线:
扩增:使用含有dUTP的引物扩增每个特定的TALE基本单位的编码基因;
切除dUTP:使用USER酶混合物切除dUTP,在每个基本单位的编码基因DNA两端保留一段特定的粘性末端;
组装:这些粘性末端按照预先设计的顺序排列,以确保所有基本单位按顺序组装连接成最终靶向特定序列的TALE工具。
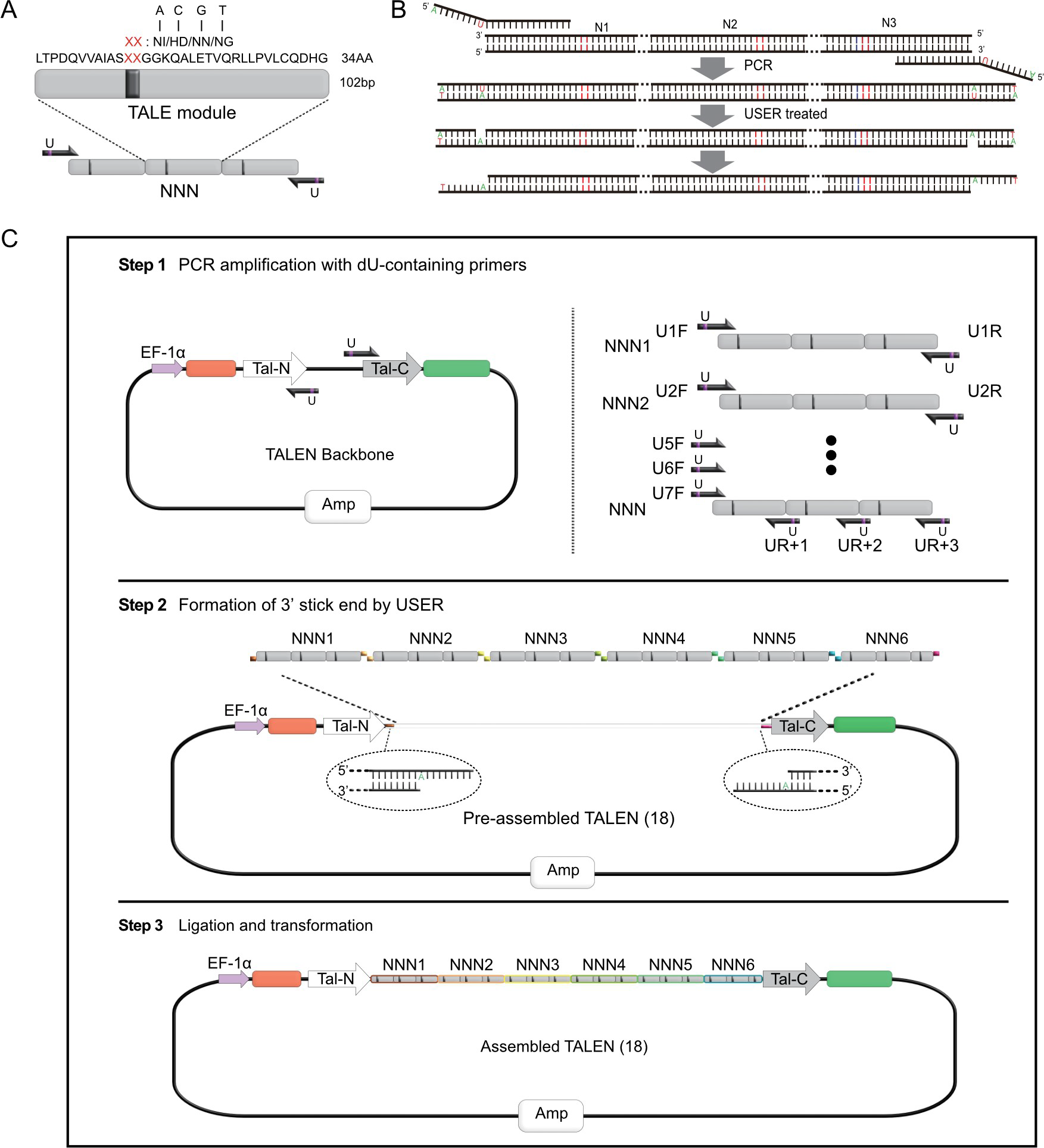
图4. 基于尿嘧啶切除克隆的TALE组装示意图[3]
助力HIT-scISOseq技术,将单细胞多个cDNA串联建库
技术路线:
获得单细胞cDNA:借助10X Genomics 平台,获得单细胞全长cDNA;
扩增cDNA:利用生物素化的含dUTP的PCR引物对全长cDNA进行扩增;
捕获cDNA:然后使用链霉亲和素珠捕获扩增的生物素化的cDNA;
切除dUTP:使用USER酶混合物切除dUTP,在cDNA的两个末端产生粘性末端;
连接与测序:使用DNA连接酶连接多个cDNA构建CCS文库后进行长读长测序。
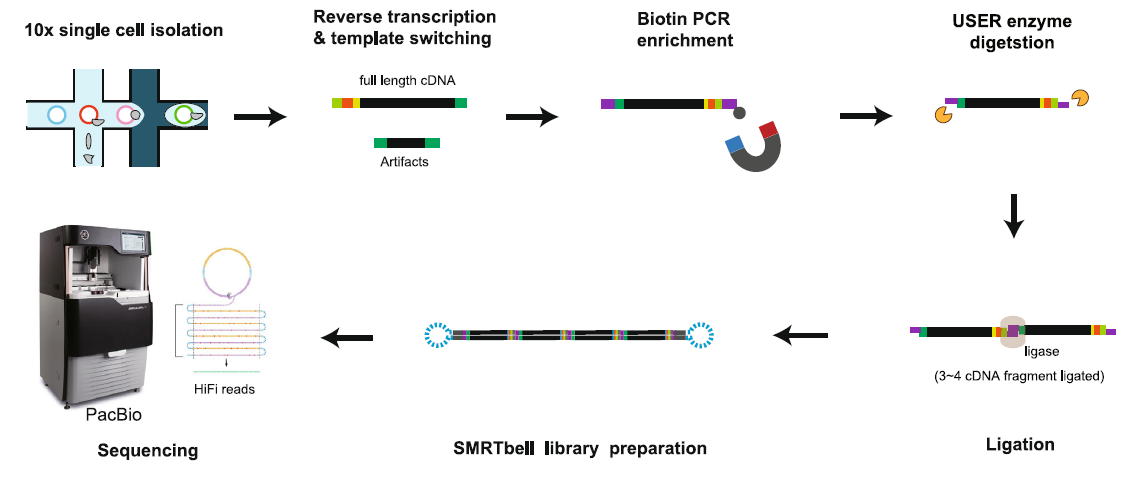
图5. HIT-scISOseq单细胞全长转录组数据的技术流程[4]
翌圣Uracil-Specific Excision Reagent
数据展示
dUTP切除效果与进口品牌N*一致
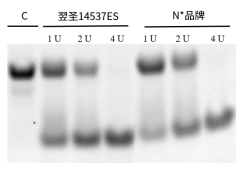
图6. dUTP切除效果对比图
USER-LIC效果优于进口品牌N*
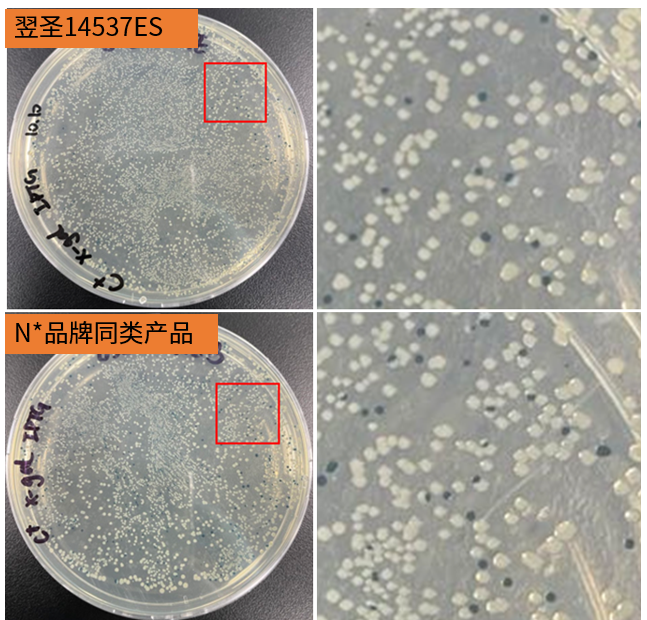
图7. 菌落数对比
客户反馈
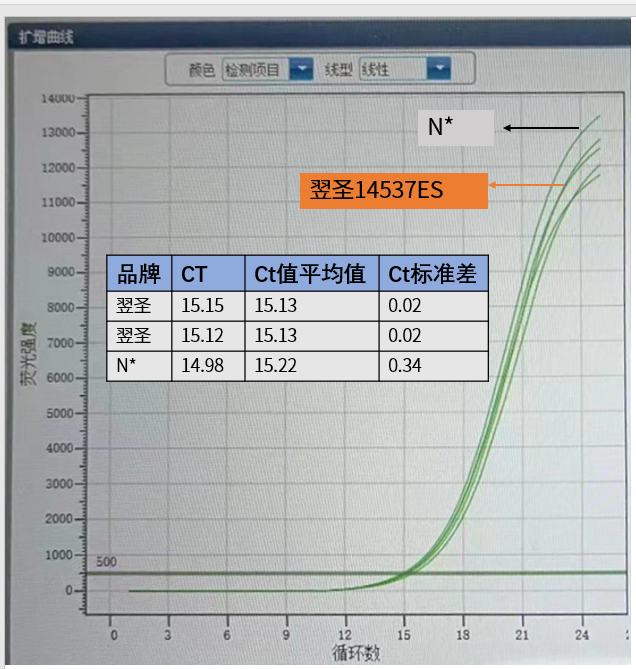
图8. qPCR扩增曲线
超低的批间差异和高剪切效率
客户测试翌圣3个批次的Uracil-Specific Excision Reagent的剪切效率,柱状图表明翌圣Uracil-Specific Excision Reagent超低的批间差异和高剪切效率。
图9. 不同批次间的剪切效率
相关产品推荐
|
应用 |
产品名称 |
产品货号 |
|
dUTP切除,USER-LIC,RNA链特异性建库 |
Uracil-Specific Excision Reagent(1 U/μL) |
|
|
DNA损伤修复研究 |
Endonuclease VIII (10 U/μL) |
|
|
PCR防污染 |
Uracil DNA Glycosylase (UDG/UNG), 1 U/μL |
参考文献
