涨知识|克隆专题二:不同分子克隆方法介绍(下)
克隆是英文“clone”或“cloning”的音译,而英文“clone”则起源于希腊文“Klone”,原意是指以幼苗或嫩枝插条。1963年J.B.S.Haldane在题为“人类种族在未来二万年的生物可能性”的演讲上采用“克隆(Clone)”的术语,把“克隆技术”带到了人们的视野中。
克隆技术又称为“生物放大技术”,目前应用最广泛的技术为“分子克隆”,又称重组DNA技术,即通过重组技术将目的DNA片段按照人们的设计定向连接起来,在特定的受体细胞中与载体同时复制并得到表达,产生新的遗传性状的技术。
之前小翌给小伙伴们介绍了常规的分子克隆方法,接下来继续带领大家认识一下新的、非常规的分子克隆技术,包括无缝克隆中的Golden Gate技术、Gateway技术、不依赖基因序列和连接反应的克隆方法等等。
Golden Gate克隆技术通过TypeIIS型限制性内切酶(翌圣BsmBI Cat#15203ES、BspQI限制性内切酶Cat#15202ES、FuniCut™ BsaI Cat#15005ES)识别目的序列以外的四个碱基,剪切后获得粘性末端,直接用于目的片段的连接。通过连接酶(翌圣T4 DNA连接酶Cat#10300)连接形成重组质粒,使用转化技术转化至感受态细胞中,由于限制性内切酶的特性,酶切连接后酶切位点不再存在,能达到精确的无缝克隆。Engler等人利用该技术成功连接了9个目的片段,连接率达90%以上。
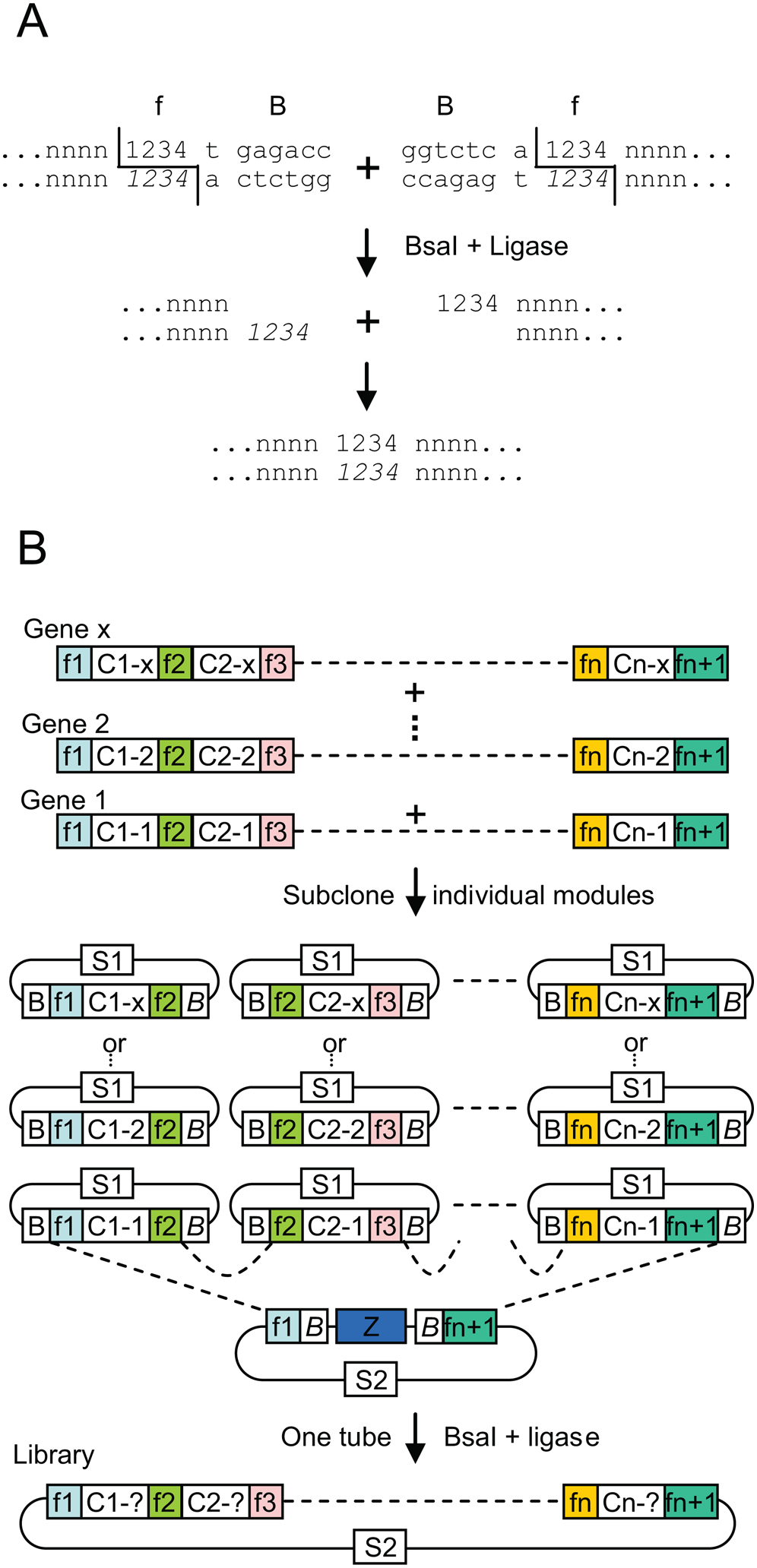
图1. Golden Gate Assembly克隆原理示意图【1】
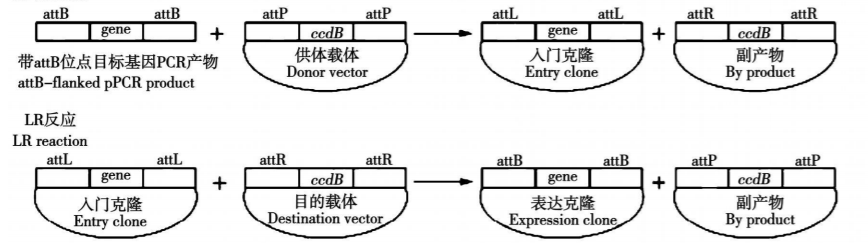
图2. Gateway技术构建表达载体【2】
基本操作流程如下:

图3. SLIC法构建表达载体的步骤【3】
其具体的操作步骤是,利用PCR技术在目的片段两侧分别加上20-25 bp的同源序列,用DpnI酶(翌圣FuniCut™ DpnI Cat#15052ES)消化处理,组装片段按合适的比例混匀,加入大肠杆菌细胞裂解物,37℃反应1 h,然后直接转化大肠杆菌感受态细胞,即可实现体外无痕组装DNA。
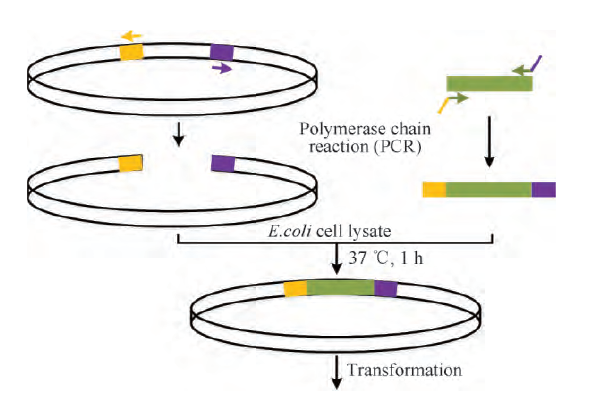
图4. SLiCE技术【4】
|
产品定位 |
产品名称 |
产品货号 |
规格 |
|
Golden Gate组装 |
BspQI限制性内切酶 (10 U/μL) |
500 U |
|
|
Golden Gate组装 |
BsmBI(10 U/μL) |
1000 U |
|
|
通用缓冲液,5min完成精准酶切 |
FuniCut™快速限制性内切酶 |
50 T |
|
|
T4连接 |
Hieff® Gold T4 DNA Ligase |
1000 U |
|
|
5 min快速完成感受态转化 |
F DH5α化学感受态 |
10×100 μL |
|
|
常规化学感受态 |
DH5α 化学感受态细胞 |
10×100 μL |
|
|
常规化学感受态 |
TOP10化学感受态细胞 |
10×100 μL |
|
|
常规PCR |
2×Hieff® PCR Master Mix(With Dye) |
1 mL/5×1 mL |
|
|
常规PCR,不含染料 |
2×Hieff® PCR Master Mix(No Dye) |
1 mL/5×1 mL |
|
|
快速PCR,快至1s/kb |
2×Hieff® Ultra-Rapid HotStart PCR Master Mix(with Dye) |
1 mL/5×1 mL |
2.林春晶, 韦正乙, 蔡勤安等. 几种植物转基因表达载体的构建方法[J].生物技术, 2008(05): 84-87.
3.张阳璞, 杨淑慎. 几种新型植物基因表达载体的构建方法[J].生物工程学报, 2015, 31(03): 311-327.
4.杨发誉, 杨云彭, 霍毅欣. 合成生物学中不依赖限制性内切酶的分子克隆策略[J].中国生物化学与分子生物学报, 2018, 34(04): 364-370.
