不同SNP检测技术的原理——ARMS、KASP、TaqMan、分子信标及HRM
SNP检测方法众多,大约有20多种,包括基于阵列的杂交、PCR和测序等,本文主要介绍基于PCR技术的部分基因分型方法。
ARMS PCR的全称是突变扩增阻滞系统PCR(Amplification Refractory Mutation System PCR),也称为等位基因特异性PCR(Allele-Specific PCR, AS-PCR),是基于Taq DNA聚合酶无法修复引物3’末端的单个碱基错配,从而使得扩增受阻的检测方法。
ARMS PCR将SNP位点设计在上游引物3’末端,结合Taqman探针的方法检测荧光信号值,从而判断野生型和突变型等位基因。换言之,我们应用ARMS PCR对野生型和突变型等位基因进行检测时,需要设计两条3’端核苷酸分别对野生型和突变型特异的上游引物,以及一条通用的下游引物,一条通用的探针。在Taq DNA聚合酶作用下,与模板不能完全匹配的上游引物将不能完全形成互补碱基对,从而产生错配,链延伸反应因3’,5’-磷酸二酯键形成障碍而受阻,不能进行延伸,而与模板完全匹配的引物体系则可以持续延伸,由于Taq酶5’到3’外切酶活性,可将Taqman探针上的荧光基团水解,导致探针上荧光基团与淬灭基团的距离增大,从而使得荧光信号被仪器检测到,最终计算出对应的△Ct值。
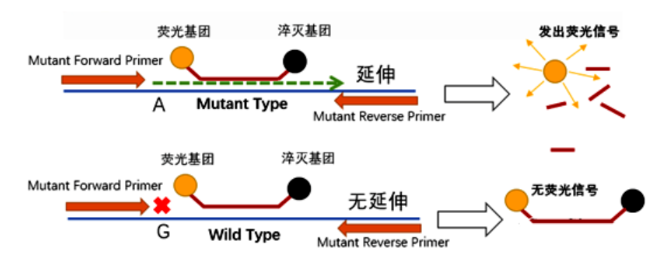
图2.ARMS-PCR检测原理
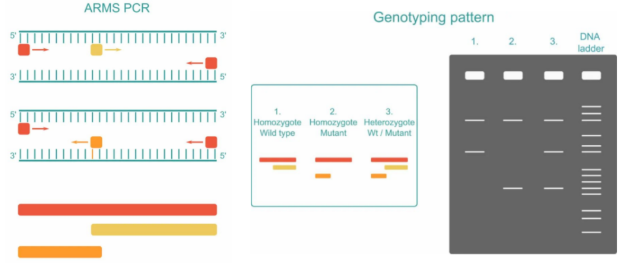
图3.四引物扩增阻滞突变系统PCR检测原理
引物探针设计
首先,针对SNP的等位基因设计两条对应的上游引物,引物3’末端分别位于各自的SNP位点上,同时引物5’端各自带有一段独特的标签序列,而下游引物则是一条常规设计共用的引物。引物一共是三条,两条带有标签序列的分型上游及一条通用下游。其次,设计两条与上游引物标签序列一致的荧光探针,探针的5’端分别标记不同的荧光基团,同时对应于荧光探针设计各自互补配对的淬灭探针,并在淬灭探针的3’端标记淬灭基团,探针一共是四条,两条荧光探针和两条淬灭探针。
在第1轮PCR扩增中,等位基因特异引物(3'末端能配对的)就可识别特定等位基因模板,完成等位基因识别,反向通用引物也会结合并完成整个PCR过程,在此阶段,荧光探针仍与其互补的淬灭探针结合,不会产生荧光信号。从第2轮PCR扩增开始,产物中出现含有通用标签序列(tag sequence)的模板,这步之后即可把通用标签序列引入与SNP对应的PCR产物中。随后荧光探针与tag互补的DNA链结合而添加到PCR产物中,因此不再与其互补的淬灭探针结合,从而产生荧光信号而被检测到。
利用荧光信号检测仪或荧光定量PCR仪(配有相应荧光探针的检测通道)进行信号读取,并用软件采集信号判别等位基因类型(如果进行聚类分析,要用到KlusterCaller或SNPviewer软件)。
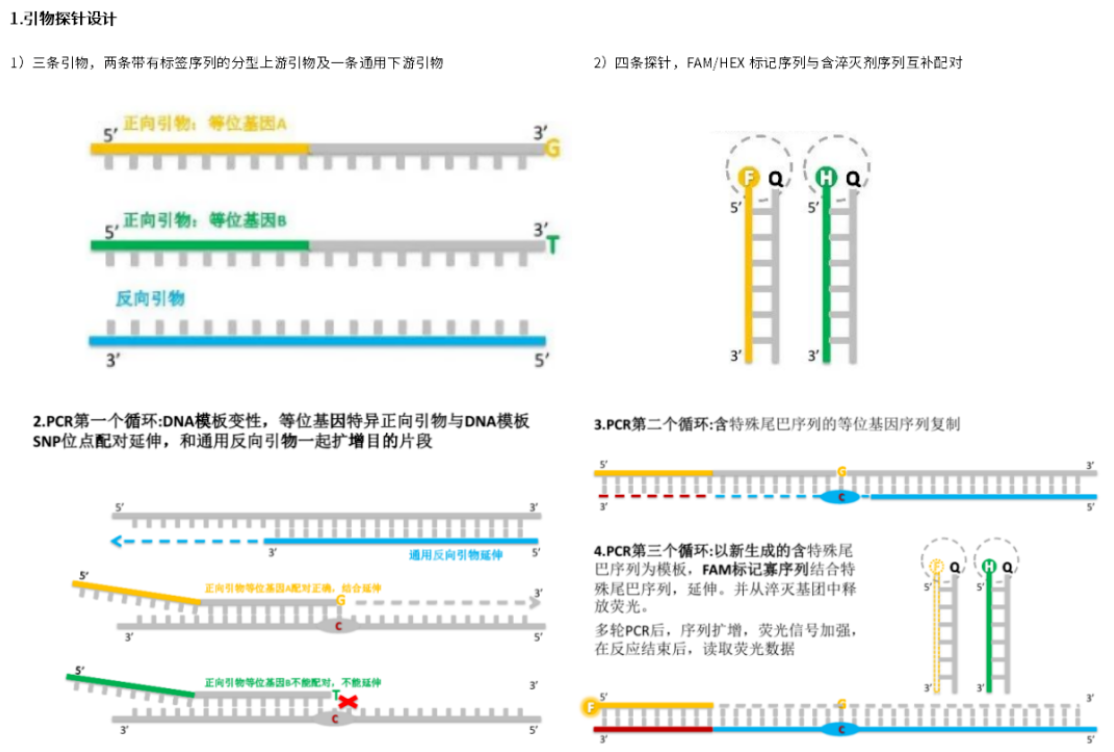
图4.KASP法检测原理
TaqMan探针是一种双标记、自淬灭的水解探针,在PCR反应中加入两条两端带有不同荧光标记的特异探针来识别不同的等位基因。其5’和3’末端分别标记荧光基团和淬灭基团,在探针结构完整时两者距离较近,荧光基团的信号可被淬灭。而在PCR扩增过程中,若TaqMan探针与靶标序列完全匹配,探针则可结合在DNA模板上,此时Taq酶延伸至探针位置时,其外切酶活性将切割水解探针,释放荧光基团,使得荧光信号增强。而且,随着扩增产物的增多,荧光信号越来越强,从而可通过仪器实时监测荧光信号的变化过程。
由于MGB探针的长度可以设计得更短(短至13个碱基),有利于提高探针的识别特异性,因此用于检测SNP的TaqMan探针常用MGB修饰,在TaqMan探针的3’端结合小沟结合物(minor groove binder,MGB),MGB与DNA螺旋的小沟(minor groove)契合,通过稳定DNA双螺旋结构来提高杂交的准确性和稳定性。另外MGB探针包括一个不发光的淬灭基团(NFQ),能够真正消除传统淬灭基团产生的背景荧光信号,提高信噪比,提高检测灵敏度。
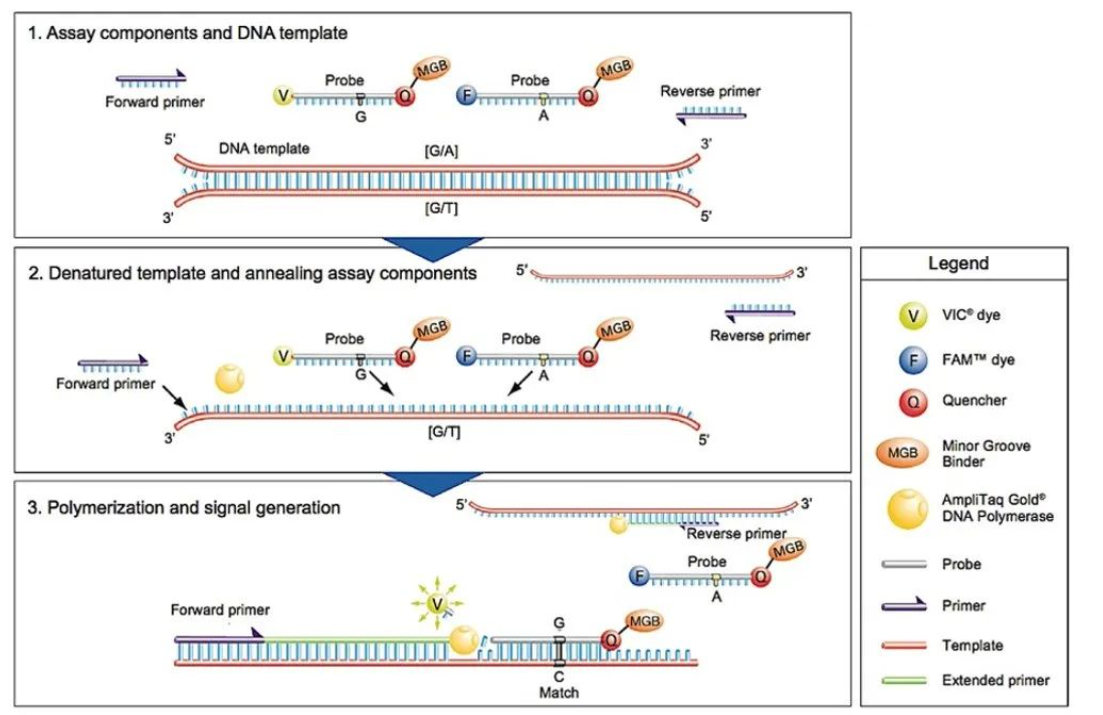
图5 TaqMan探针法检测原理
分子信标是一段双标记的寡核苷酸探针,其5’末端和3’末端分别标记荧光基团和淬灭基团,且探针5’端和3’端的部分碱基可互补配对,从而形成茎环结构,使得荧光基团和淬灭基团相互靠近而荧光信号较低。分子信标的环状结构部分包含SNP检测位点,当模板与分子信标环状结构的核酸序列完全匹配时,分子信标可与模板杂交形成伸展状态,从而导致荧光基团和淬灭基团的空间距离拉大,荧光信号增强,因此可被仪器检测,并确定SNP位点。使用不同荧光基团标记的分子信标,则可区分SNP类型。
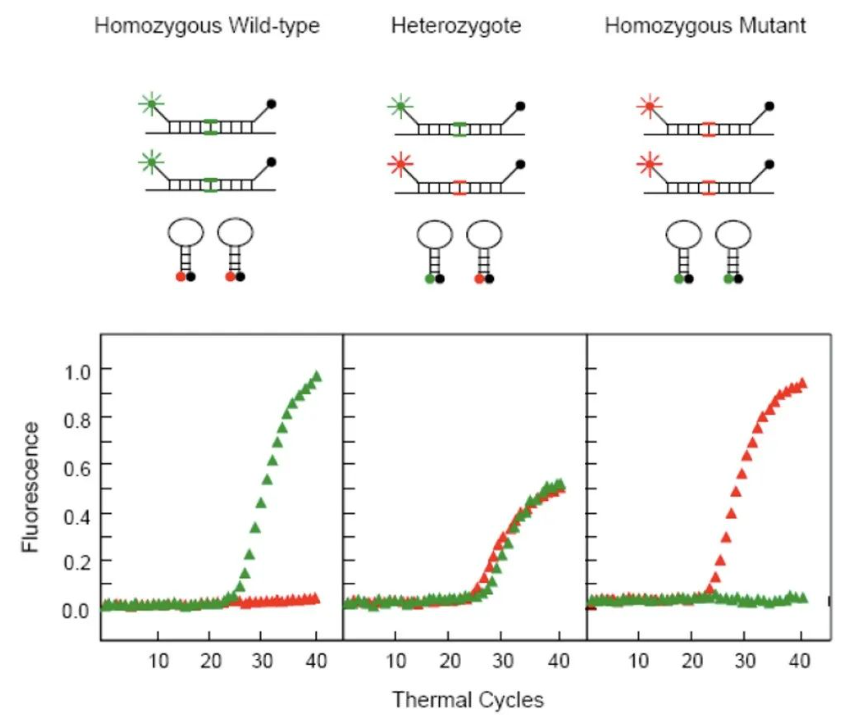
图6.分子信标法检测原理
分子信标法与TaqMan探针法类似,均是通过探针中单个碱基的识别能力区分SNP,只是分子信标采用茎环结构,而TaqMan探针使用MGB修饰,以提高探针对SNP的识别特异性。该方法本底低,特异性高,灵敏性高,不必与未反应的探针分离即可检测,可用于活体分析;但是探针合成时标记较复杂,设计难度大。在进行分子信标的研究和应用时,茎干区和环状区的序列设计是关键所在,这种设计不仅决定了其构象,使在杂交后淬灭基团与荧光基团之间的距离达到足够大,而且决定了其合适的使用温度。其次,环境pH值、纯度也是影响分子信标的重要因素,pH值过高,分子信标的发卡结构可能被破坏,出现假阳性结果。
PCR扩增的熔解曲线取决于其扩增序列,序列中一个碱基的突变都可以导致双链DNA的解链温度发生变化,通过实时荧光定量PCR仪监测这种细微的温度变化,可以知道扩增的序列中是否有突变发生,从而对其进行基因分型。
高分辨率熔解曲线(high-resolution melting analysis, HRM)是通过特定染料与扩增特定的DNA区域,在高分辨率熔解条件下对扩增产物进行温度升降,通过熔解曲线的变化区分不同的基因型或等位基因。HRM检测通常使用饱和荧光染料(如:LC Green、LC Green Plus、SYTO 9等),具有更强的DNA结合能力,且不影响PCR扩增,在DNA解链过程中也不会发生重排,使得熔解曲线具有更高的分辨率。
核酸片段的固有特性,如DNA序列的长度、GC含量和碱基互补性差异等,均能影响高分辨率熔解曲线,而结合高精度荧光定量PCR仪,其分辨精度则可达到对单个碱基差异的区分,从而可用于确定SNP位点。
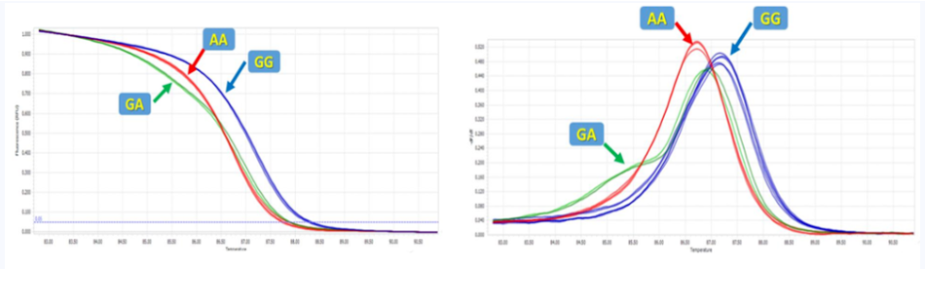
图7.高分辨率熔解曲线法检测SNP结果示意图
该方法引物设计简单,且单碱基改变、插入、缺失都能通过HRM来检测,实验操作简单,不需要探针,成本相对较低,并且可以发现未知突变(但不能知道具体的突变类型)。但是HRM技术对实验仪器的温度分辨率要求相当高,需要达到0.02~0.1℃,每升高1℃采集荧光25次,以满足对单碱基差异的区分。对温度均一性同样要求较高,Tm的标准偏差为0.020-0.264℃(孔间温度均一性要达到0.264℃以内才能保证HRM分析结果的准确性),一般PCR两孔间温差在0.3~0.5℃,这就导致两孔间最终熔解温度相差较大,无法保证HRM分析结果的准确性。
翌圣生物作为上游原料企业,在分子酶领域深耕多年,目前已开发了ARMS-PCR法及TaqMan探针法的SNP分型检测通用原料,已被下游厂家应用于肿瘤伴随诊断、药物基因组学、单基因遗传病、疾病易感性研究等多个领域。
| 产品名称 | 货号 | 备注 |
| 2×Hieff Unicon® TaqMan SNP Genotyping Master Mix | 13152ES | TaqMan探针法 |
| 2×Hieff® Blood Direct TaqMan qPCR Master Mix(Low ROX) | 13095ES | |
| 2×Hieff Unicon® Multiplex ARMS qPCR Mix | 13755ES | ARMS-PCR法 |
| 2×Hieff Unicon® Multiplex ARMS qPCR Kit | 13229ES | |
| Hieff UNICON® HotStart Super Specific Taq DNA Polymerase | 14318ES | 单酶 |
| 10×Taq PCR Buffer(with MgCl2) | 11373ES | 通用Buffer |
通过上面SNP科普般的分享,相信大家对部分主流SNP检测方法及特点有了基本的了解,那SNP主要在哪些应用领域起作用,以及常见的SNP问题有哪些,限于篇幅原因,敬请期待下篇SNP干货分享哦!
