一文全面解析NGS接头的奥秘
NGS测序技术近年来得到了巨大发展,2017年摩根大会上,Illumina重磅推出NovaSeq测序仪,NovaSeq可以2天产生2Tb数据,通量是10年前Genome Analyzer的2000倍。测序通量的大幅增加,意味着更多的样本混合上机,这样的话我们如何在茫茫数据中找到对应样本匹配的数据呢?人们想到了一种在文库构建时在接头上添加“接头暗号”的方法,在测序完成之后根据“接头暗号”对样本进行分离。这里的接头暗号就是样本标签“Index/Barcode”。本文将以建库过程中要添加的接头为核心,对其进行全面介绍。
1.接头是什么?
接头的本质是一段短的碱基序列,基本包括三个部分:与flow-cell上面寡核苷酸相同或互补的片段P5/P7;测序时测序引物结合部分R1/R2;用于区分不同样本的Index。接头是待测DNA片段与Flow-cell连接的桥梁,目的片段连接接头后可以在flow cell上扩增再测序。

图1.两端添加接头的DNA片段
2.接头如何分类?
接头的分类方法主要有两种,一是按照Index的位置,二是按照是否匹配PCR free建库。
(1)根据Index位置可以将接头分为单端Index接头和双端Index接头。单端Index接头指的是仅在P5端或P7端存在Index(一般在P7端),双端Index接头指的在P5和P7端均存在Index。(如图2所示)。Index的数目直接影响最终上机能混合的样本数目,双端Index具有比单端Index能容纳更多数目的样本,近年来为了满足一次能测量更多的样本的需求,双端带Index的接头被广泛使用。
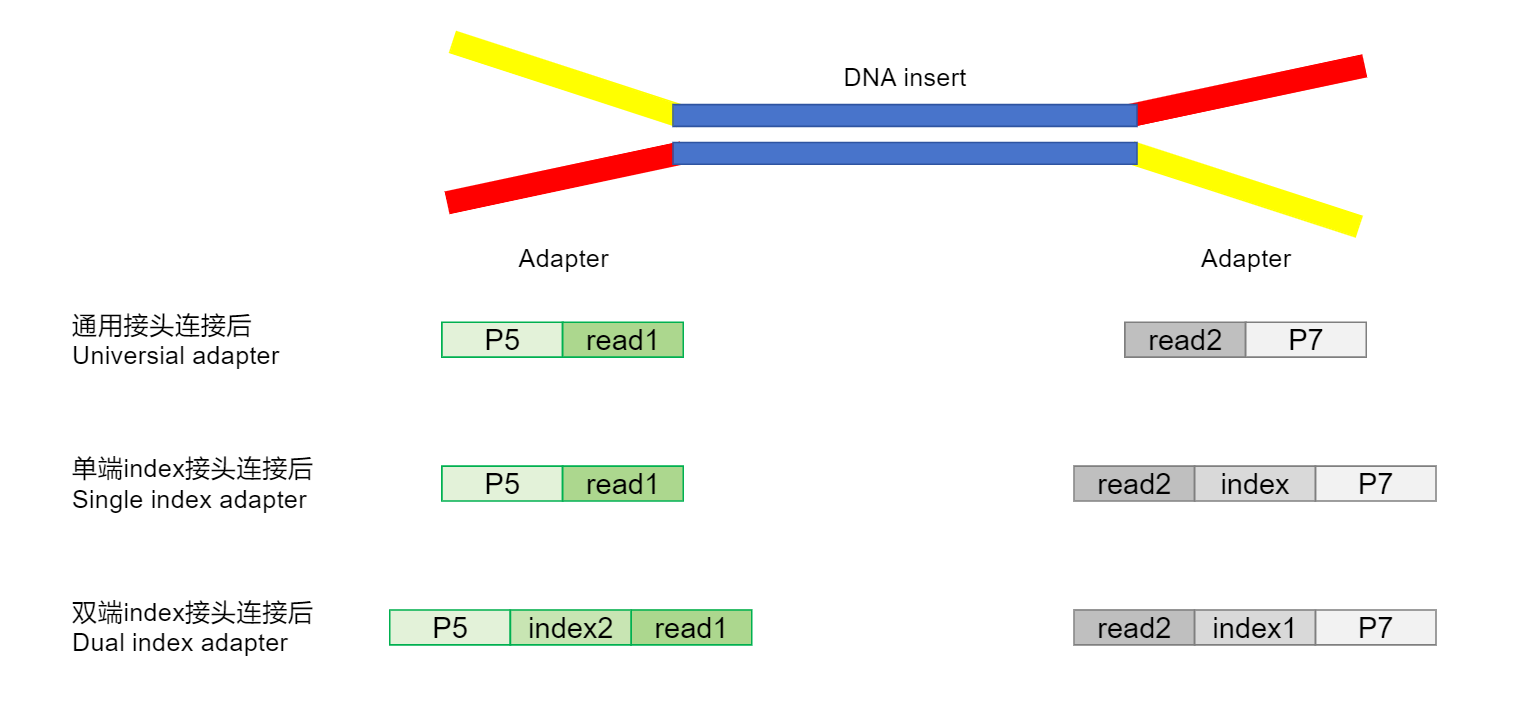
图2:接头按照Index位置分为单端Index接头和双端Index接头,两种接头连接后示意图
(2)根据接头是否匹配PCR free建库可以将接头分为长接头和短接头(见图3)。长接头又称为完整接头,包括P5/P7+Index序列+Read 1/2,完整接头通过TA克隆的方式连接到DNA片段之后,可不进行PCR扩增反应直接上机测序(DNA量足够上机测序时可直接上机,当DNA量不够时还需进行PCR扩增使得产物达到一定的量方可上机测序)。短接头通过TA克隆方式连接到DNA片段上后,必须与短接头互补的引物进行PCR扩增,扩增产物就是包含完整接头的DNA片段(见图4)。也就是说短接头最终一定要通过PCR扩增成为完整接头才能上机测序。
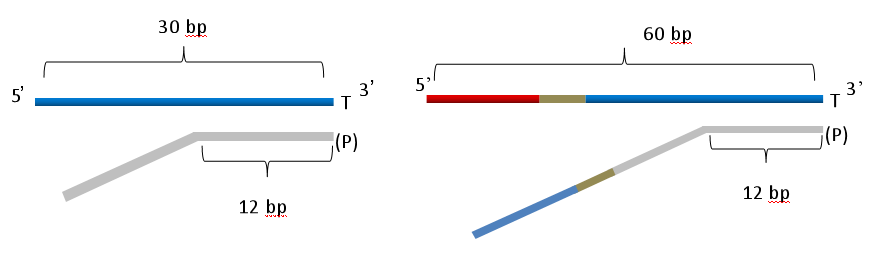
图3:左边为Illumina早期推出的短接头,不包含Index;右边为长接头,是Illumina目前标准通用接头样式
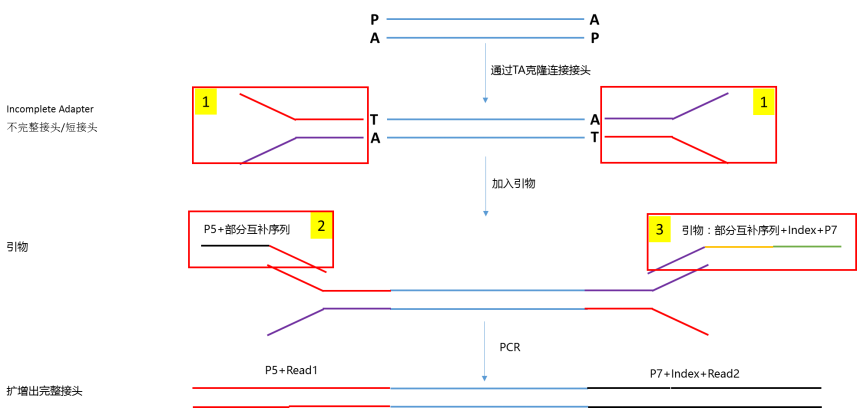
图4:短接头建库流程图
3.Index有何奥秘?
Index作为接头中的重要组成部分到底有着怎样的奥秘呢?简单来说Index就是混合样本中不同样本的“身份证”,其本身就是一段碱基序列,一般长6nt或8nt。通过对这种“身份证”的识别,就可以在混合样本中对单个样本的数据进行识别。那么问题来了,四种碱基随机构成的排列组合序列那么多,这些都可以用作Index吗?选择Index序列的依据又是什么呢?
Index的选择需满足两个原则:碱基平衡和激光平衡
a)碱基平衡:是指Index序列的复杂度和平衡度:复杂度指的是碱基的种类的多样;平衡度指的是碱基之间分布比例的均衡。需要注意的是碱基的平衡是指多个Index之间的平衡,而不是单个index内部的碱基平衡。好的Index序列应该是均含有A、T、C、G四种碱基,且各碱基之间的比例接近25%,如图5所示。
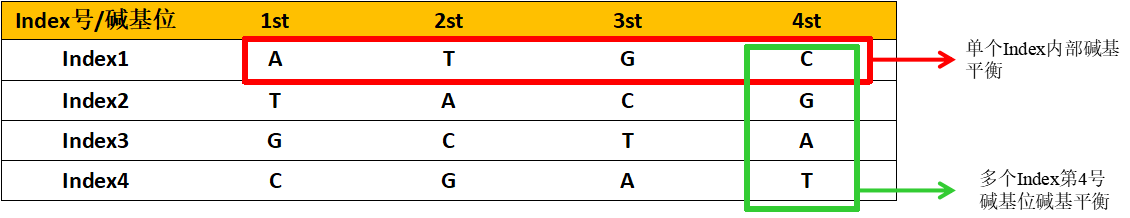
图5:符合碱基平衡的Index号(ATGC,TACG,GCTA,CGAT)举例,分别符合单个Index内部平衡和多个Index相应碱基位的碱基平衡
b)激光平衡:是指在一组Index序列中需满足每个碱基位A + C =G + T,在Illumina测序仪中,A和C两种碱基共用一种激光,由波长660nm的红激光激发;G和T共用一种激光,由波长532 nm的绿激光激发。需要说明的是激光平衡是在碱基不平衡的情况下的无奈之举,在一定程度上可以提高index测序时的碱基识别质量,减少数据分离时出问题的可能性,见图6。
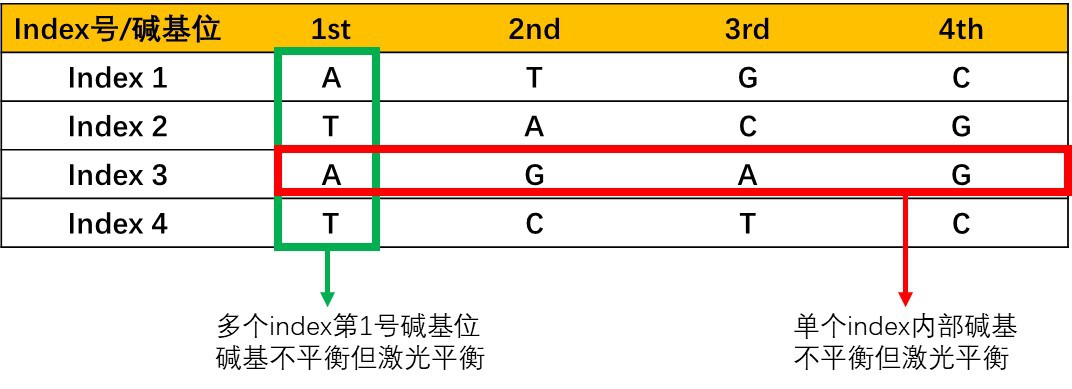
图6:不符合碱基平衡但符合激光平衡的Index号(ATGC,TACG,AGAG,TCTC)举例
如样本数为单数,则必然无法满足碱基平衡和激光平衡,此时可以选择几个纵列是完全互补的两个Index,再加上一个其他的Index,可最大程度保证测序质量。
结语
高通量测序技术的发展,使得测序的通量不断增加,通量的增加就意味着多样本混合上机测序。由此而来的问题是在NGS的过程中如何做到对每个样本数据的对号入座。科学家想到给样本一个“身份证”,这样就可以凭证识别。因此对于NGS从业者来说,在享受高通量测序带来的方便快捷的同时,还要记得给自己的样本戴好‘证件’哦。
