原核生物转录组研究优势,看这篇!
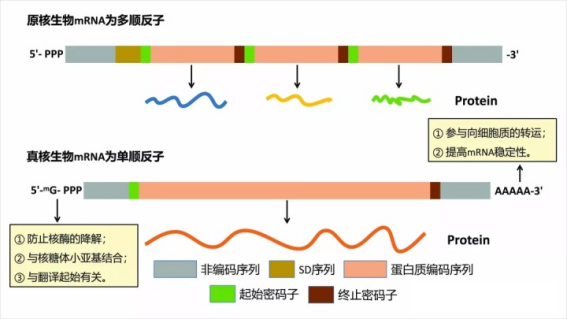
基因结构注释更准确
原核生物基因组测序后,一般通过软件分析注释基因编码区及非编码RNA。软件注释一来有可能出错,而且一些非编码区域检测不出来或者UTR区域注释不到。原核生物转录组能够鉴定出一些保守的小肽(有可能在基因组注释分析时被过滤掉)。
mRNA的非编码调控区域的检测及注释
原核生物mRNA的非编码区域含有重要的调控元件如核糖开关和小RNA结合位点等。核糖开关指的是mRNA一些非翻译区的序列折叠成一定的构象,这些构象的改变应答于体内的一些代谢分子,从而通过这些构象的改变达到调节mRNA转录的目的。但是核糖开关通过基因组测序分析是没有办法鉴定,只有在原核生物转录组的不同培养条件的差异分析才能被鉴定到。
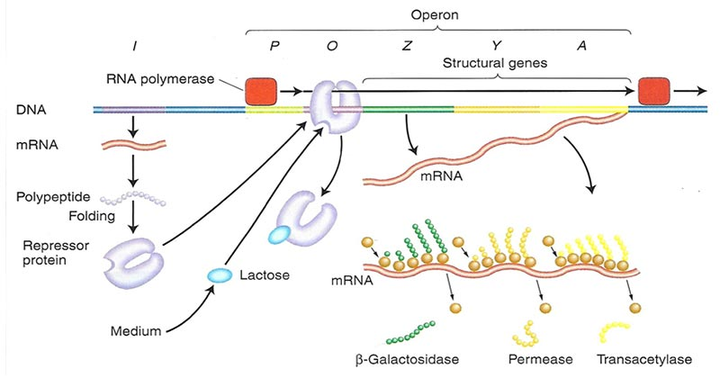
操纵子结构功能
原核生物基因组中的基因通常排列在操纵子中,一个多反式mRNA包含几个共转录的基因。但是有文献报道[1],在不同条件下操纵子结构调控机制不一样,即在一种条件下编码在多顺反子中的基因可以在另一种条件下转录为单顺反子转录物。原核生物转录组研究有利于利用基于转录组的实验工具来确定操纵子结构,有望推动我们对原核生物多顺反子转录物的多用途调控和进化的理解。
非编码RNA调控研究
sRNA在原核生物生理调控中起重要作用,这些sRNAs通常在50到500 bp之间,已被证明可以调节重要的生物过程,如毒力、应激反应和群体感应等。大多数特征的sRNAs通过与目标5’UTR的碱基配对来调节其mRNA靶标的翻译或稳定性,在功能上类似于真核miRNAs。原核生物sRNA测序有助于更好地了解原核生物sRNA的功能。
RNA元件功能的可塑性
原核生物转录组研究,能让我们了解到RNA元件在不同的条件下,行使功能不一样。如L. monocytogenes菌[2]中,赖氨酸存在的情况下,赖氨酸核糖开关形成转录终止子,因此下游转运蛋白不表达。但是在赖氨酸缺失的情况下,核糖开关的再折叠允许赖氨酸转运基因的转录。
|
产品名称 |
货号 |
规格 |
|
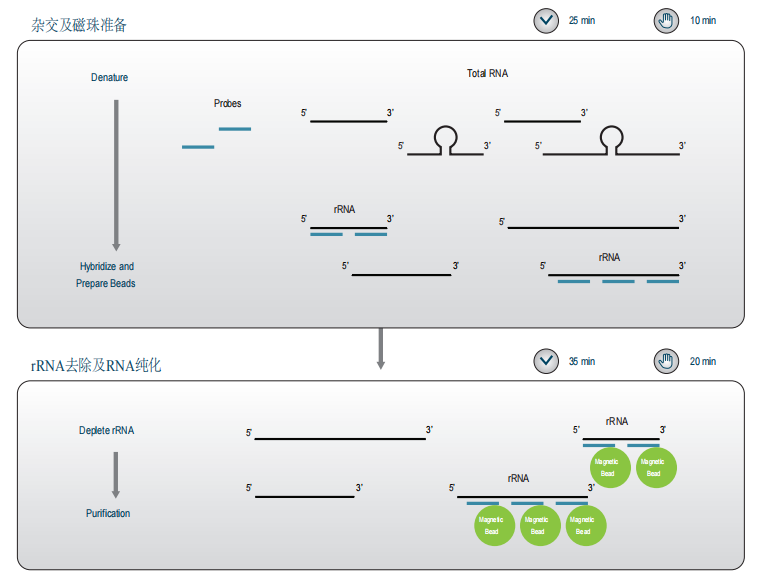
Hieff NGS® MagSP rRNA Removal Kit (Bacteria(G- and G+)) with purification beads |
12264ES24/96 |
24 T/ 96 T |
|
Hieff NGS® MagSP rRNA Removal Kit (Prokaryote) with purification beads |
12265ES24/96 |
24 T/ 96 T |
|
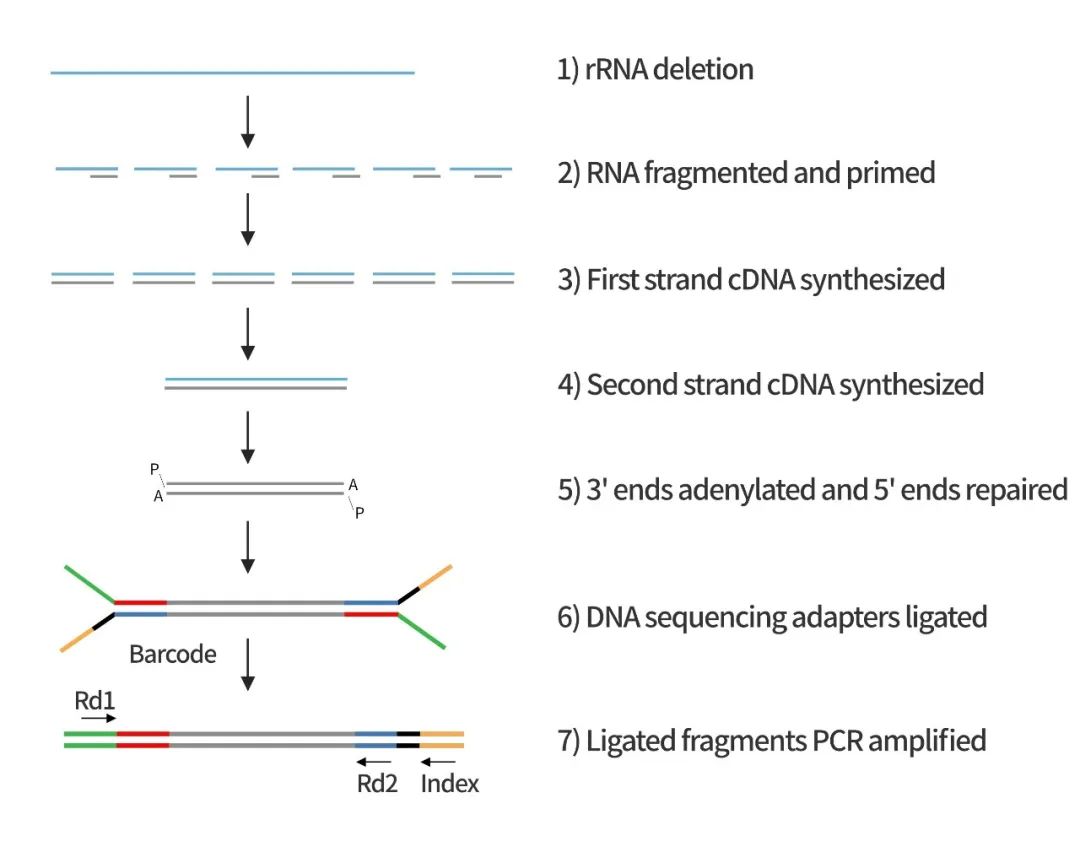
Hieff NGS® Ultima Dual-mode RNA Library Prep Kit(Premixed version) |
12310ES24/96 |
24 T/ 96 T |
