带你揭开low input甲基化建库的神秘面纱
DNA甲基化在真核生物基因组表观遗传调控中起着关键作用,因此,5-甲基胞嘧啶(或称甲基组)在全基因组分布一直备受关注。全基因组亚硫酸氢盐测序(WGBS)是一种既能获得单碱基分辨率又能获得全基因组覆盖的方法,已成功应用于真核系统发育树各分支,多个物种的甲基组的分析,以及人类胚胎干细胞、诱导多能干细胞、外周血单个核细胞、结肠癌细胞等甲基组的分析中。这些WGBS数据给我们带来了很多其他方法无法获得的新发现,随着测序成本的降低,WGBS越来越成为研究的首选方法。然而传统WGBS方法对低投入量(low input)样本来说有较大的困难,随着甲基化应用场景的不断增加,无论从胚胎发育的研究到肿瘤及早筛的临床应用,低投入量样本甲基化建库的需求越来越多。
虽然WGBS已经被证明是强大的技术,但传统的WGBS方法是先进行文库构建,到连接接头后再进行亚硫酸盐转化,有一些实际的局限性:
首先,WGBS需要的样本投入量比较高,WGBS的主要挑战是在亚硫酸氢盐转化,亚硫酸盐转化过程是一个具有较大破坏性的化学转化过程。DNA在完全转化的条件下会发生严重的降解。通常,约90%投入的DNA会被降解,连接好接头的文库,再经过亚硫酸盐处理,断裂掉的分子则不能会后续扩增出来,从而损失掉。因此WGBS通常需要ug级的DNA作为起始材料。从而许多生物学上一些有意思的样本(如早期胚胎、胚胎组织和哺乳动物的卵子以及cfDNA)的文库制备就比较困难,甚至会失败。那么当样本量有限时,这种方法并不可用。
此外,富含未甲基胞嘧啶的区域对链断裂更敏感。经亚硫酸盐处理后,非甲基化的胞嘧啶转化为尿嘧啶,DNA中这些转化的区域更容易发生断裂,断裂后的片段因两端不含有完整的接头,从而不是一个有效的文库分子,这些模板被排除掉,可能会导致不完全覆盖和一定的偏向性。
最后,对于WGBS文库制备效率低的原因,有研究表明,亚硫酸氢盐处理试剂盒制备的DNA质量收率并没有那么低,从30%到70%不等。因此,WGBS文库制备中产量低的最主要原因不是亚硫酸氢盐处理的回收率,而是接头连接和亚硫酸盐处理的顺序。亚硫酸氢盐引起的DNA降解导致完整的文库分子急剧减少,这是文库制备效率低的主要原因。因此,为了解决这一问题,一种先转化后建库的方法Post-Bisulfite Adaptor Tagging (PBAT)被提出。
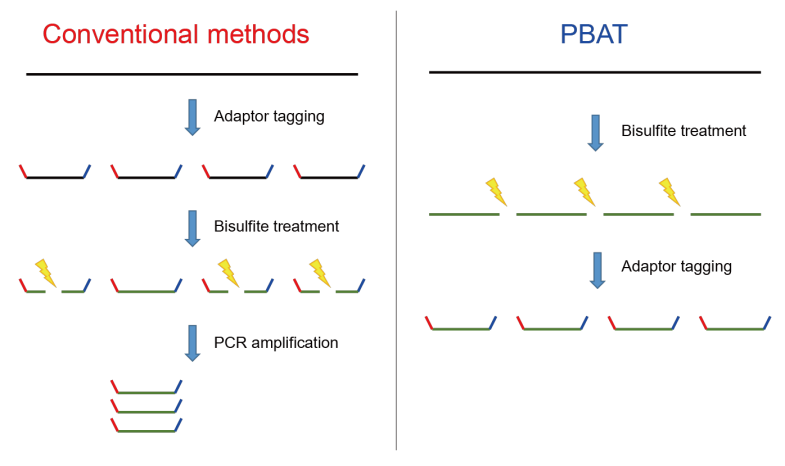
图1.PBAT可以高效地制备全基因组亚硫酸氢盐测序文库(Nucleic Acids Research, 2019, Vol. 47, No. 15)
那么先转化再建库怎么实现呢?由于亚硫酸盐处理的DNA是单链的,PBAT的实现需要一种有效的方法将接头序列连接到单链DNA (ssDNA)上。研究者们需要在单链DNA上做研究,一种通过两轮随机引物扩增,被称为随机启动介导的PBAT,即random priming-mediated PBAT (rPBAT)的方法被开发出来。图2所示经过亚硫酸盐处理后的样本通过两轮随机引物扩增,连接上完整的接头,即为完整的文库。
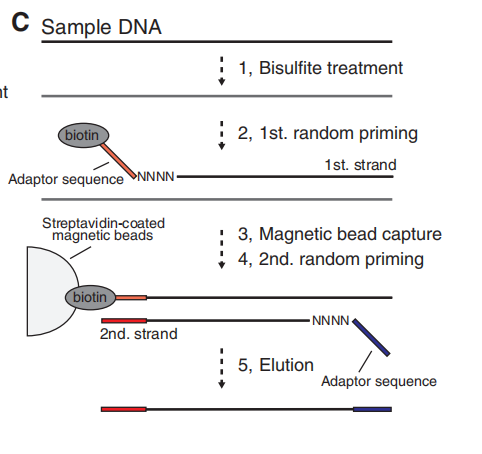
图2.随机引物介导的PBAT方法(Nucleic Acids Research, 2012, Vol. 40, No. 17)
随机启动介导的PBAT使得甲基化文库构建的效率大大提高,甚至可能从125 pg基因组DNA中制备WGBS文库。通过不断地优化,rPBAT甚至被用于单细胞甲基组测序,尽管基因组覆盖率有所降低。事实上,rPBAT文库,特别是那些从极少量DNA (250 pg)中制备的文库,包含越来越多的不能Mapping的reads,已经表明,rPBAT文库包含相当大比例的嵌合读长。rPBAT的这些缺点可能归因于随机引物启动反应。
首先,随机启动通常不是从3'端开始,而是从目标ssDNA片段内的一个位点开始,从而留下一个边缘序列,该边缘序列不包括在新合成的待测序的reads中。因此,每个库片段不可避免地比它的目标ssDNA短,特别是当使用两轮随机引物反应时。第二,随机引物的效率取决于随机引物-基因组DNA双链的稳定性。因此,随机启动反应通常会导致对GC丰富区域的偏好,而对AT丰富区域的基因组覆盖效率较低。第三,随机引物不仅发生在引物和基因组DNA之间,也发生在两个引物之间,以及两个基因组DNA片段之间,从而分别产生不可Mapping和嵌合的reads。
因此,为了解决这个问题,可以通过消除任意一个或两个随机引物扩增的步骤,来延长库片段的长度、减少GC偏向覆盖、减少不可Mapping和嵌合率来进一步改善rPBAT。
虽然rPBAT制备文库的效率要远远高于传统WGBS方法,但rPBAT仍仅将约10%的投入DNA转化为文库片段,仍有进一步改进的空间。那么有没有能减少随机引物扩增步骤的方法呢?研究者发现有两种可以替代随机引物扩增法进行接头连接。一种是使用RNA连接酶,它可以将5'端磷酸化的接头连接到3'末端。但是RNA连接酶对DNA oligo之间的连接效率要远低于RNA之间的连接。另外一种方法是通过末端脱氧核糖核酸转移酶(TdT)介导的末端加尾法连接到ssDNA的3'端,末端序列可做为引物,合成ssDNA的互补链。有研究者将两种方法组合了一种叫Tdt辅助连接的PBAT (tPBAT)的方法,该方法也成功的从125 pg的人类基因组DNA中制备了tPBAT文库。
前面提到,由于随机引物很少与目标ssDNA的3'端杂交,不易覆盖亚硫酸盐转化DNA片段全长的DNA。而且,两轮随机引物扩增使得到的DNA片段短得多。tPBAT法通过Tdt连接取代了传统rPBAT方法中的第二个随机引物步骤,产生了更多插入片段更长的文库分子。图3结果表明tPBAT法确实能够解决原来rPBAT法插入片段短的问题。
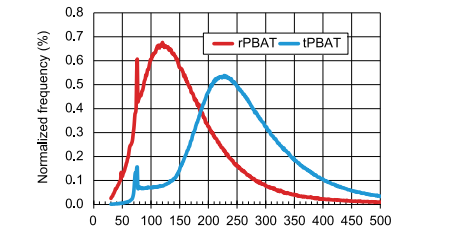
图3.两种PBAT方法mapping 到基因组上reads长度分布(Nucleic Acids Research, 2019, Vol. 47, No. 15)
对于tPBAT不同投入量的研究表明(图4),其具有和rPBAT几乎相同的灵敏性,即均能对低投入量样本进行甲基化建库。但当投入DNA的起始量小于1ng时,rPBAT文库的分子产量超过了tPBAT文库的分子产量(图4)。不过,投入和产出呈近似线性关系。表1中测序结果发现tPBAT文库产量在低投入量时要低,但mapping率要远高于rPBAT法。随着投入DNA的减少,rPBAT和tPBAT文库的Mapping率分别下降。分析其原因,tPBAT和rPBAT的净产出上在很大程度上是可比较的,因为tPBAT在库片段长度上优于rPBAT,另外嵌合体也会占据一定比例的文库产量。
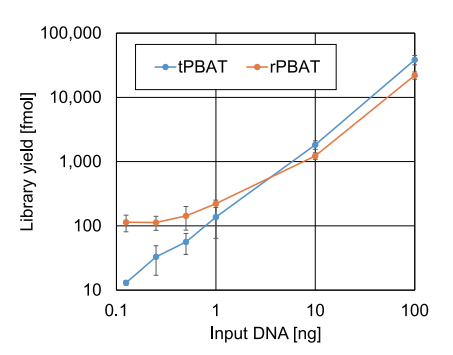
图4.tPBAT和rPBAT法DNA投入量和文产出的关系(Nucleic Acids Research, 2019, Vol. 47, No. 15)
表1.tPBAT和rPBAT的文库收率和reads Mapping率对比(Nucleic Acids Research, 2019, Vol. 47, No. 15

除了上述的方法之外,还有一些更简单高效的针对亚硫酸盐处理的甲基化文库构建方法,应用于少量或者降解样本。随着技术的发展,更有效、更少偏差和更长片段的WGBS文库构建方法将不断涌现出来,对于低投入量的PBAT法也加速WGBS的应用,得以在各种有限制的样品中使用,甚至包括博物馆样品和濒危物种的组织,这些物种的DNA通常严重降解甚至变性或者样本量比较少。
今天就先分享到这里,更多干货请持续关注翌生生物公众号的更新。翌生有一款针对低投入量(1-200ng DNA)的甲基化建库试剂盒,更多信息欢迎咨询。
[1] Miura F, Enomoto Y, Dairiki R, Ito T.Amplification-free whole-genome bisulfite sequencing by post-bisulfite adaptor tagging. Nucleic Acids Res. 2012 Sep 1;40(17):e136.
[2] Khanna, A., Czyz, A. & Syed, F. EpiGnome™ Methyl-Seq Kit: a novel post–bisulfte conversion library prep method for methylation analysis. Nature Methods
[3] Miura F, Shibata Y, Miura M, Sangatsuda Y, Hisano O, Araki H, Ito T.Highly efficient single-stranded DNA ligation technique improves low-input whole-genome bisulfite sequencing by post-bisulfite adaptor tagging. Nucleic Acids Res. 2019 Sep 5;47(15):e85.
[4] Miura,F. and Ito,T. (2015) Highly sensitive targeted methylome sequencing by post-bisulfite adaptor tagging. DNA Res., 22, 13–18
