Hieff NGS® Ultima Pro DNA Library Prep Kit是针对Illumina®和MGI®高通量测序平台专业开发设计的新一代建库试剂盒。本产品采用高质量的酶学组成,在前一代建库试剂盒的基础上,改进了DNA片段末端修复和加A效率,同时改善了接头连接效率。本试剂盒采用新型的高保真酶,显著提高扩增均一性和保真性,可应用于常规动植物基因组、微生物基因组、FFPE、cfDNA、ChIP DNA等样本,助力获得优异的测序数据。
- 适用100pg-1000ng所有DNA样本,包含cfDNA、FFPE等。
- 业界领先的文库转化率,最高可达70%以上。
- 多样本验证可获得优异的文库与测序数据。
- 严格的批次性能与稳定性质控。
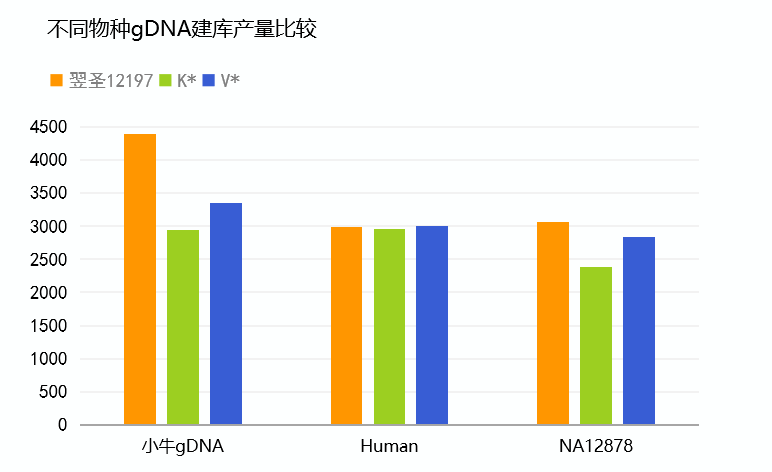
与竞品试剂盒相比,12197建库产量相对较高
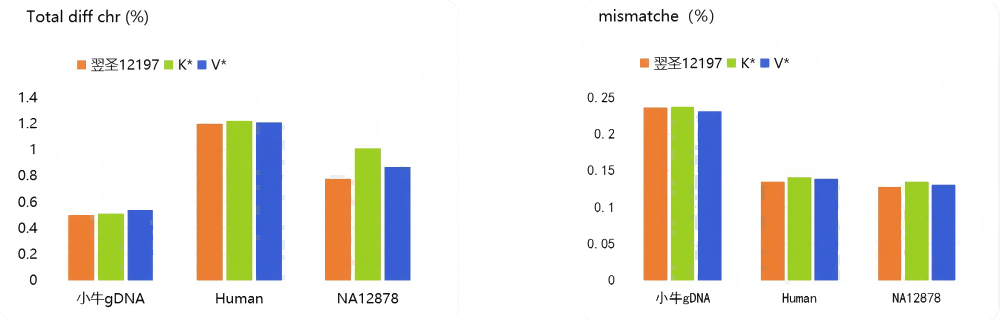
与竞品试剂盒相比,12197下机数据,片段自连较低、mismatch较低。
-25~-15℃保存。
[1] Cha N, Jia B, He Y, et al. MicroRNA-124 suppresses the invasion and proliferation of breast cancer cells by targeting TFAP4. Oncol Lett. 2021;21(4):271. doi:10.3892/ol.2021.12532(IF:3.111)
[2] Ma W, Zhang X, Liu Y. miR-124 promotes apoptosis and inhibits the proliferation of vessel endothelial cells through P38/MAPK and PI3K/AKT pathways, making it a potential mechanism of vessel endothelial injury in acute myocardial infarction. Exp Ther Med. 2021;22(6):1383. doi:10.3892/etm.2021.10819(IF:2.751)